Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Orientation of Biotin-Binding Sites in Streptavidin Adsorbed onto the Surface of Polythiophene Films
Langmuir ( IF 3.7 ) Pub Date : 2019-01-30 00:00:00 , DOI: 10.1021/acs.langmuir.8b03509 Kamil Awsiuk 1, 2 , Panagiota Petrou 2 , Angelos Thanassoulas 2 , Joanna Raczkowska 1
Langmuir ( IF 3.7 ) Pub Date : 2019-01-30 00:00:00 , DOI: 10.1021/acs.langmuir.8b03509 Kamil Awsiuk 1, 2 , Panagiota Petrou 2 , Angelos Thanassoulas 2 , Joanna Raczkowska 1
Affiliation
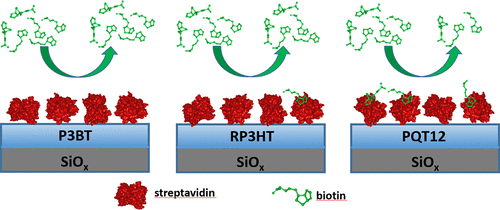
|
The orientation of biotin-binding sites of streptavidin adsorbed to thin films of three polythiophenes (PTs), namely, regioregular poly(3-hexylthiophene) (RP3HT), regiorandom poly(3-butylthiophene) (P3BT), and poly(3,3‴-didodecylquaterthiophene) (PQT12), has been investigated. Polymer films were examined prior to and after protein adsorption with atomic force microscopy and time-of-flight secondary ion mass spectrometry (ToF-SIMS). Principal component analysis (PCA) applied to ToF-SIMS data revealed subtle changes in surface chemistry of polymer films and orientation of adsorbed streptavidin. PCA resolved the surface alignment of alkyl side chains and differentiated the ToF-SIMS data for PQT12, RP3HT, and P3BT, verifying an amorphous morphology for P3BT and a semicrystalline one for PQT12 and RP3HT. After the characterization of the polymeric films, streptavidin adsorption from solutions with different protein concentrations (up to 300 μg/mL) has been conducted. The PCA results distinguished between amino acids characteristic for external regions of streptavidin molecules adsorbed to different PTs suggest that streptavidin adsorbed to PQT12 exposes molecular regions rich in tryptophan and tyrosine, which are components of the biotin-binding sites. The latter results were confirmed using biotin-labeled horse radish peroxidase to estimate the exposed binding sites of streptavidin adsorbed onto the different PT films. The analysis of streptavidin structure suggests that interaction between polythiophene film and dipole moment of streptavidin subunit is responsible for orientation of biotin-binding sites.
中文翻译:

链霉亲和素吸附到聚噻吩薄膜表面上的生物素结合位点的方向
链霉亲和素的生物素结合位点吸附到三种聚噻吩(PT)的薄膜上的取向,这三种聚噻吩是区域规则的聚(3-己基噻吩)(RP3HT),区域无规的聚(3-丁基噻吩)(P3BT)和聚(3,3)已研究了β-二十二烷基季四噻吩(PQT12)。在蛋白质吸附之前和之后,通过原子力显微镜和飞行时间二次离子质谱(ToF-SIMS)检查聚合物薄膜。应用于ToF-SIMS数据的主成分分析(PCA)显示了聚合物膜表面化学性质和吸附的链霉亲和素取向的细微变化。PCA解析了烷基侧链的表面排列并区分了PQT12,RP3HT和P3BT的ToF-SIMS数据,从而验证了P3BT的非晶态和PQT12和RP3HT的半晶态。表征聚合物膜后,已从具有不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了链霉亲和素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。已经从不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了抗生蛋白链菌素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。已经从不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了抗生蛋白链菌素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。
更新日期:2019-01-30
中文翻译:

链霉亲和素吸附到聚噻吩薄膜表面上的生物素结合位点的方向
链霉亲和素的生物素结合位点吸附到三种聚噻吩(PT)的薄膜上的取向,这三种聚噻吩是区域规则的聚(3-己基噻吩)(RP3HT),区域无规的聚(3-丁基噻吩)(P3BT)和聚(3,3)已研究了β-二十二烷基季四噻吩(PQT12)。在蛋白质吸附之前和之后,通过原子力显微镜和飞行时间二次离子质谱(ToF-SIMS)检查聚合物薄膜。应用于ToF-SIMS数据的主成分分析(PCA)显示了聚合物膜表面化学性质和吸附的链霉亲和素取向的细微变化。PCA解析了烷基侧链的表面排列并区分了PQT12,RP3HT和P3BT的ToF-SIMS数据,从而验证了P3BT的非晶态和PQT12和RP3HT的半晶态。表征聚合物膜后,已从具有不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了链霉亲和素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。已经从不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了抗生蛋白链菌素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。已经从不同蛋白质浓度(最高300μg/ mL)的溶液中吸附了抗生蛋白链菌素。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。PCA结果区分了链霉亲和素分子吸附到不同PT的外部区域的特征性氨基酸,这表明吸附到PQT12的链霉亲和素暴露了富含色氨酸和酪氨酸的分子区域,色氨酸和酪氨酸是生物素结合位点的组成部分。使用生物素标记的辣根过氧化物酶来评估吸附在不同PT膜上的链霉亲和素的暴露结合位点,证实了后者的结果。对链霉亲和素结构的分析表明,聚噻吩膜与链霉亲和素亚基的偶极矩之间的相互作用是生物素结合位点定向的原因。































 京公网安备 11010802027423号
京公网安备 11010802027423号