当前位置:
X-MOL 学术
›
ACS Chem. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
通过区域特异性突变和选择对Caspase-7特异性进行重编程,为底物识别提供了替代解决方案
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2016-03-31 00:00:00 , DOI: 10.1021/acschembio.5b00971 Maureen E. Hill 1 , Derek J. MacPherson 1 , Peng Wu 1 , Olivier Julien , James A. Wells , Jeanne A. Hardy 1
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2016-03-31 00:00:00 , DOI: 10.1021/acschembio.5b00971 Maureen E. Hill 1 , Derek J. MacPherson 1 , Peng Wu 1 , Olivier Julien , James A. Wells , Jeanne A. Hardy 1
Affiliation
|
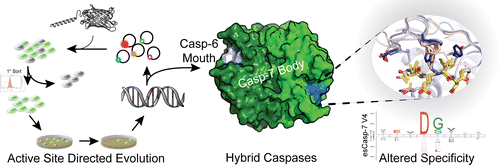
常规工程化蛋白酶特异性的能力可以使我们更好地了解和调节其生物学特性,以扩展治疗和工业应用。在这里,我们报告了一种基于笼状绿色荧光蛋白(CA-GFP)报告基因的新方法,该方法允许在细菌或其他细胞类型中基于流式细胞仪进行选择,从而能够选择胞内蛋白酶特异性,而无需考虑蛋白酶的组成复杂性。在这里,我们应用这种方法将caspase-6的特异性引入caspase-7,caspase-7是一种对细胞重塑和细胞死亡很重要的细胞内半胱氨酸蛋白酶。我们发现将caspase-6中与底物接触的残基替换为caspase-7无效,从而产生了失活的酶,而在这些位置的饱和诱变和通过定向进化的选择产生了活跃的胱天蛋白酶。该过程产生了许多非显而易见的突变,这些突变使得caspase-7特异性能够与caspase-6匹配。进化特异性caspase-7(esCasp-7)的结构揭示了底物的替代结合模式,包括重组活性位点环。通过以下方法对esCasp-7的整个人类蛋白质组进行分析N-末端经济学表明,对天然蛋白质底物的整体特异性与caspase-6非常相似。因为esCasp-7保持了caspase-7的核心,所以我们能够鉴定caspase-6底物,lamin C,我们预测它依赖于外源来识别底物。这些重新编程的蛋白酶可能是第一个以区分异位点依赖性或非依赖性底物的明确意图构建的工具。这种特异性重编程的方法也应该在广泛的蛋白酶中推广。

"点击查看英文标题和摘要"
更新日期:2016-03-31

"点击查看英文标题和摘要"






































 京公网安备 11010802027423号
京公网安备 11010802027423号