当前位置:
X-MOL 学术
›
Nat. Protoc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Efficient and robust proteome-wide approaches for cross-linking mass spectrometry.
Nature Protocols ( IF 13.1 ) Pub Date : 2018-Dec-01 , DOI: 10.1038/s41596-018-0074-x Oleg Klykov , Barbara Steigenberger , Sibel Pektaş , Domenico Fasci , Albert J. R. Heck , Richard A. Scheltema
Nature Protocols ( IF 13.1 ) Pub Date : 2018-Dec-01 , DOI: 10.1038/s41596-018-0074-x Oleg Klykov , Barbara Steigenberger , Sibel Pektaş , Domenico Fasci , Albert J. R. Heck , Richard A. Scheltema
|
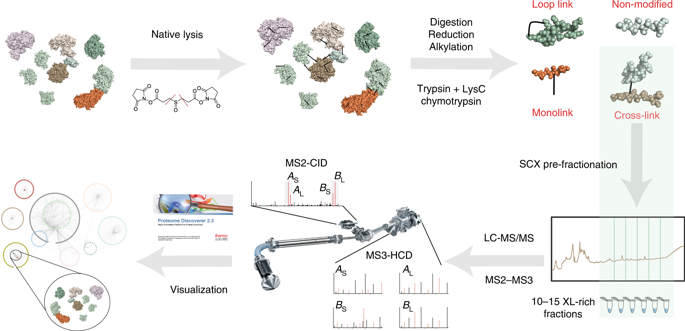
Cross-linking mass spectrometry (XL-MS) has received considerable interest, owing to its potential to investigate protein-protein interactions (PPIs) in an unbiased fashion in complex protein mixtures. Recent developments have enabled the detection of thousands of PPIs from a single experiment. A unique strength of XL-MS, in comparison with other methods for determining PPIs, is that it provides direct spatial information for the detected interactions. This is accomplished by the use of bifunctional cross-linking molecules that link two amino acids in close proximity with a covalent bond. Upon proteolytic digestion, this results in two newly linked peptides, which are identifiable by MS. XL-MS has received the required boost to tackle more-complex samples with recent advances in cross-linking chemistry with MS-cleavable or reporter-based cross-linkers and faster, more sensitive and more versatile MS platforms. This protocol provides a detailed description of our optimized conditions for a full-proteome native protein preparation followed by cross-linking using the gas-phase cleavable cross-linking reagent disuccinimidyl sulfoxide (DSSO). Following cross-linking, we demonstrate extensive sample fractionation and substantially simplified data analysis with XlinkX in Proteome Discoverer, as well as subsequent protein structure investigations with DisVis and HADDOCK. This protocol produces data of high confidence and can be performed within ~10 d, including structural investigations.
中文翻译:

高效,强大的蛋白质组学方法,用于交联质谱。
交联质谱法(XL-MS)备受关注,因为它有潜力以无偏见的方式研究复杂蛋白质混合物中的蛋白质-蛋白质相互作用(PPI)。最近的发展使从单个实验中检测成千上万的PPI成为可能。与其他确定PPI的方法相比,XL-MS的独特优势在于,它可为检测到的相互作用提供直接的空间信息。这是通过使用双功能交联分子来实现的,该分子将两个氨基酸与共价键紧密相连。通过蛋白水解消化,产生两个新连接的肽,其可通过MS鉴定。XL-MS通过使用MS可裂解或基于报告分子的交联剂以及更快,更灵敏和更通用的MS平台,在交联化学领域取得了最新进展,从而获得了解决复杂样品所需的动力。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。更灵敏,功能更广泛的MS平台。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。更灵敏,功能更广泛的MS平台。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。
更新日期:2019-01-26
中文翻译:

高效,强大的蛋白质组学方法,用于交联质谱。
交联质谱法(XL-MS)备受关注,因为它有潜力以无偏见的方式研究复杂蛋白质混合物中的蛋白质-蛋白质相互作用(PPI)。最近的发展使从单个实验中检测成千上万的PPI成为可能。与其他确定PPI的方法相比,XL-MS的独特优势在于,它可为检测到的相互作用提供直接的空间信息。这是通过使用双功能交联分子来实现的,该分子将两个氨基酸与共价键紧密相连。通过蛋白水解消化,产生两个新连接的肽,其可通过MS鉴定。XL-MS通过使用MS可裂解或基于报告分子的交联剂以及更快,更灵敏和更通用的MS平台,在交联化学领域取得了最新进展,从而获得了解决复杂样品所需的动力。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。更灵敏,功能更广泛的MS平台。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。更灵敏,功能更广泛的MS平台。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。该协议详细介绍了我们用于全蛋白质组天然蛋白制备的优化条件,然后使用气相可裂解的交联剂二琥珀酰亚胺基亚砜(DSSO)进行交联。交联后,我们证明了在Proteome Discoverer中使用XlinkX进行广泛的样品分离和实质上简化的数据分析,以及随后使用DisVis和HADDOCK进行的蛋白质结构研究。该协议可产生高置信度的数据,可在〜10 d内执行,包括结构研究。































 京公网安备 11010802027423号
京公网安备 11010802027423号