当前位置:
X-MOL 学术
›
Nano Lett.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
核酸组装的多链结构预测和 RNA 开关的设计
Nano Letters ( IF 9.6 ) Pub Date : 2016-02-29 00:00:00 , DOI: 10.1021/acs.nanolett.5b04651 Eckart Bindewald 1 , Kirill A Afonin 2, 3 , Mathias Viard 1 , Paul Zakrevsky 2 , Taejin Kim 2 , Bruce A Shapiro 2
Affiliation
|
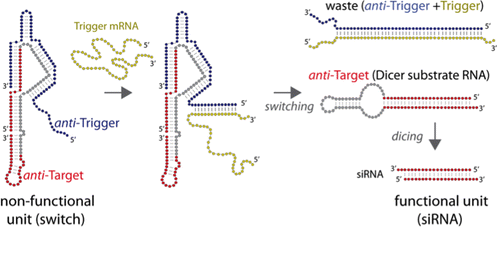
RNA 是创建分子逻辑门的一种有吸引力的材料,这种分子逻辑门仅在特定分子相互作用伙伴存在的情况下才释放编程功能。在这里,我们介绍了 HyperFold,一种多链 RNA/DNA 结构预测方法,用于预测可能包含假结的核酸复合物。我们表明,与其他已发布的折叠算法相比,HyperFold 的性能也具有竞争力。我们针对不同浓度、DNA 立足点长度和 G+C 含量进行了大量的 RNA/DNA 杂合重关联实验,发现观察到的重关联趋势与计算预测吻合良好。重要的是,我们将此方法应用于双链RNA分子开关的设计和实验验证,该开关在与单链RNA立足点疾病标记物触发mRNA结合后改变其构象,释放类似shRNA的Dicer底物结构。为了证明这一概念,分别选择结缔组织生长因子 (CTGF) mRNA 和增强型绿色荧光蛋白 (eGFP) mRNA 作为触发序列和目标序列。体外实验证实了RNA开关的形成,并证明当触发RNA与开关立足点相互作用时,功能单元被释放。设计的 RNA 开关在 MDA-MB-231 乳腺癌细胞中显示出功能。还设计并测试了其他几种开关。我们的结论是,这种方法具有相当大的潜力,因为原则上,它允许释放针对与用作疾病状态生物标志物的基因不同的基因设计的 siRNA。

"点击查看英文标题和摘要"




















































 京公网安备 11010802027423号
京公网安备 11010802027423号