Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
通过GEO和TCGA数据的综合分析,了解肺鳞状细胞癌的转录组和功能网络特征。
Scientific Reports ( IF 3.8 ) Pub Date : 2018-Oct-26 , DOI: 10.1038/s41598-018-34160-w
Yin Li , Jie Gu , Fengkai Xu , Qiaoliang Zhu , Di Ge , Chunlai Lu
Scientific Reports ( IF 3.8 ) Pub Date : 2018-Oct-26 , DOI: 10.1038/s41598-018-34160-w
Yin Li , Jie Gu , Fengkai Xu , Qiaoliang Zhu , Di Ge , Chunlai Lu
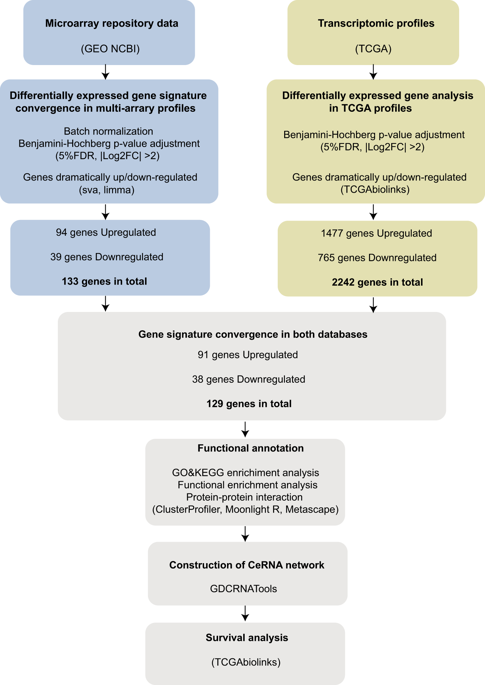
|
肺鳞状细胞癌(LUSC)与临床预后不良有关,并且缺乏可用的靶向治疗。LUSC的诊断和预后迫切需要新型分子。在这里,我们通过比较从肿瘤组织与正常组织(GSE8569,GSE21933,GSE33479,GSE33532,GSE40275,GSE62113,GSE74706)整合到基因表达综合(GEO)数据库中的差异表达基因,对LUSC进行了数据挖掘分析。基因组图谱(TCGA)数据库,其中包括502个肿瘤和49个相邻的非肿瘤肺组织。我们确定了GEO数据和TCGA数据之间的129个基因(91个上调和38个下调)的交集。基于这些基因,我们进行了下游分析,包括功能富集分析,蛋白质-蛋白质相互作用,竞争性内源RNA(ceRNA)网络和生存分析。这项研究可能通过对GEO和TCGA数据的综合分析提供对LUSC的转录组和功能特征的更多见解,并提出LUSC的治疗靶标和生物标记物。

"点击查看英文标题和摘要"
更新日期:2018-10-26

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号