当前位置:
X-MOL 学术
›
Hortic. Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Improved Brassica rapa reference genome by single-molecule sequencing and chromosome conformation capture technologies.
Horticulture Research ( IF 7.6 ) Pub Date : 2018-01-01 , DOI: 10.1038/s41438-018-0071-9 Lei Zhang , Xu Cai , Jian Wu , Min Liu , Stefan Grob , Feng Cheng , Jianli Liang , Chengcheng Cai , Zhiyuan Liu , Bo Liu , Fan Wang , Song Li , Fuyan Liu , Xuming Li , Lin Cheng , Wencai Yang , Mai-he Li , Ueli Grossniklaus , Hongkun Zheng , Xiaowu Wang
Horticulture Research ( IF 7.6 ) Pub Date : 2018-01-01 , DOI: 10.1038/s41438-018-0071-9 Lei Zhang , Xu Cai , Jian Wu , Min Liu , Stefan Grob , Feng Cheng , Jianli Liang , Chengcheng Cai , Zhiyuan Liu , Bo Liu , Fan Wang , Song Li , Fuyan Liu , Xuming Li , Lin Cheng , Wencai Yang , Mai-he Li , Ueli Grossniklaus , Hongkun Zheng , Xiaowu Wang
|
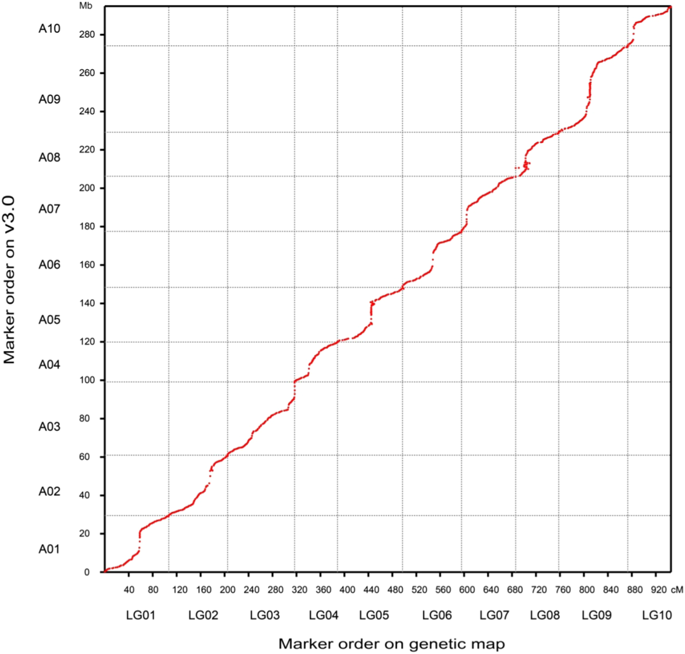
Brassica rapa comprises several important cultivated vegetables and oil crops. Current reference genome assemblies of Brassica rapa are quite fragmented and not highly contiguous, thereby limiting extensive genetic and genomic analyses. Here, we report an improved assembly of the B. rapa genome (v3.0) using single-molecule sequencing, optical mapping, and chromosome conformation capture technologies (Hi-C). Relative to the previous reference genomes, our assembly features a contig N50 size of 1.45 Mb, representing a ~30-fold improvement. We also identified a new event that occurred in the B. rapa genome ~1.2 million years ago, when a long terminal repeat retrotransposon (LTR-RT) expanded. Further analysis refined the relationship of genome blocks and accurately located the centromeres in the B. rapa genome. The B. rapa genome v3.0 will serve as an important community resource for future genetic and genomic studies in B. rapa. This resource will facilitate breeding efforts in B. rapa, as well as comparative genomic analysis with other Brassica species.
中文翻译:

通过单分子测序和染色体构象捕获技术改进的甘蓝型油菜参考基因组。
甘蓝型油菜包含几种重要的栽培蔬菜和油料作物。目前芸苔属的参考基因组装配体非常零碎并且不是高度连续的,从而限制了广泛的遗传和基因组分析。在这里,我们报告使用单分子测序,光学作图和染色体构象捕获技术(Hi-C)的B. rapa基因组(v3.0)的改进装配。相对于以前的参考基因组,我们的程序集具有1.45 Mb的重叠群N50大小,代表〜30倍的改善。我们还确定了发生在B. rapa的新事件基因组大约在120万年前,当时一个长末端重复逆转录转座子(LTR-RT)扩增了。进一步的分析完善了基因组块之间的关系,并准确定位了R. rapa基因组中的着丝粒。在白菜基因组3.0版将成为今后基因和基因组学研究的一个重要的社会资源芜菁。此资源将促进B. rapa的育种工作,以及与其他芸苔属物种进行比较基因组分析。
更新日期:2019-01-26
中文翻译:

通过单分子测序和染色体构象捕获技术改进的甘蓝型油菜参考基因组。
甘蓝型油菜包含几种重要的栽培蔬菜和油料作物。目前芸苔属的参考基因组装配体非常零碎并且不是高度连续的,从而限制了广泛的遗传和基因组分析。在这里,我们报告使用单分子测序,光学作图和染色体构象捕获技术(Hi-C)的B. rapa基因组(v3.0)的改进装配。相对于以前的参考基因组,我们的程序集具有1.45 Mb的重叠群N50大小,代表〜30倍的改善。我们还确定了发生在B. rapa的新事件基因组大约在120万年前,当时一个长末端重复逆转录转座子(LTR-RT)扩增了。进一步的分析完善了基因组块之间的关系,并准确定位了R. rapa基因组中的着丝粒。在白菜基因组3.0版将成为今后基因和基因组学研究的一个重要的社会资源芜菁。此资源将促进B. rapa的育种工作,以及与其他芸苔属物种进行比较基因组分析。































 京公网安备 11010802027423号
京公网安备 11010802027423号