当前位置:
X-MOL 学术
›
J. Phys. Chem. B
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
量子力学计算的粗粒双链RNA模型
The Journal of Physical Chemistry B ( IF 2.8 ) Pub Date : 2018-08-08 , DOI: 10.1021/acs.jpcb.8b03566 Sergio Cruz-León 1, 2 , Alvaro Vázquez-Mayagoitia 3 , Simone Melchionna 4 , Nadine Schwierz 2 , Maria Fyta 1
The Journal of Physical Chemistry B ( IF 2.8 ) Pub Date : 2018-08-08 , DOI: 10.1021/acs.jpcb.8b03566 Sergio Cruz-León 1, 2 , Alvaro Vázquez-Mayagoitia 3 , Simone Melchionna 4 , Nadine Schwierz 2 , Maria Fyta 1
Affiliation
|
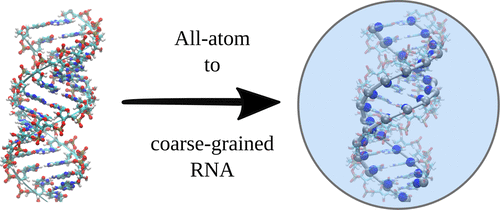
利用从量子力学计算获得的参数,开发了用于模拟双链RNA结构特性的粗粒度模型。该模型遵循先前对双链DNA的参数化,该参数化是基于将全原子图片映射到粗粒四珠方案的。RNA和DNA之间的化学和结构差异已被考虑用于模型开发。参数化基于使用密度泛函理论(DFT)对RNA分子的各个单元进行的模拟,而无需实现实验数据。总能量被分解为四个物理意义的术语:氢键相互作用,堆积相互作用,骨架相互作用和静电相互作用。前三个交互在DFT中处理,而最后一个则包含在平均场近似中。我们的双链RNA粗粒模型可预测RNA的稳定螺旋结构。其他特性(例如结构或机械特性)的复制精度很高。RNA粗粒度模型的开发允许扩展计算机仿真访问的空间和时间长度尺度,并能够对与RNA有关的生物物理过程以及新型RNA纳米结构进行建模。

"点击查看英文标题和摘要"
更新日期:2018-08-08

"点击查看英文标题和摘要"















































 京公网安备 11010802027423号
京公网安备 11010802027423号