当前位置:
X-MOL 学术
›
Mol. Catal.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
3D-QSAR辅助活性预测策略,用于扩展醛酮还原酶的底物谱
Molecular Catalysis ( IF 3.9 ) Pub Date : 2018-06-26 , DOI: 10.1016/j.mcat.2018.06.013
Qiang Cheng , Qi Chen , Jian-He Xu , Hui-Lei Yu

"点击查看英文标题和摘要"
更新日期:2018-06-26
Molecular Catalysis ( IF 3.9 ) Pub Date : 2018-06-26 , DOI: 10.1016/j.mcat.2018.06.013
Qiang Cheng , Qi Chen , Jian-He Xu , Hui-Lei Yu
|
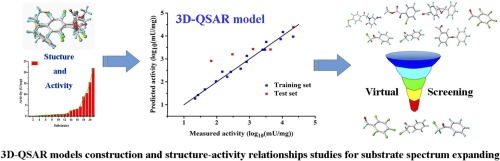
醛基酮还原酶(AKR)被广泛用于将前手性醛或酮化合物还原为相应的手性醇。在我们之前的工作中,从芽孢杆菌属中分离并鉴定了一种名为YtbE的醛酮还原酶。ECU0013,它可以以高的立体选择性催化NADPH依赖的羰基还原反应。为了进一步扩大酶的应用范围,构建了三维定量结构活性关系(3D-QSAR)模型来预测对醛或酮化合物的比活性。对于比较分子场分析(CoMFA)和比较分子相似性指标分析(CoMSIA)模型,交叉验证系数(Q 2)(0.623,0.601)是令人满意的,这表明该模型对于预测而言是鲁棒且有效的。然后通过等高线图分析检查了蛋白质YtbE与不同底物之间的详细结构-功能关系。我们发现静电和疏水力分子场在确定催化活性中起着最重要的作用,贡献因子分别为0.378和0.384。此外,使用ZINC15数据库进行了虚拟筛选,以扩展研究YtbE的底物光谱。在预测的77种潜在底物中,有5种经过实验验证,表明CoMSIA分子模型的准确性更高。事实证明,3D-QSAR辅助方法有助于合理研究和扩展YtbE底物谱。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号