当前位置:
X-MOL 学术
›
Biochemistry
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Post-Translational Modifications in Polypyrimidine Tract Binding Proteins PTBP1 and PTBP2
Biochemistry ( IF 2.9 ) Pub Date : 2018-05-31 00:00:00 , DOI: 10.1021/acs.biochem.8b00256
Jeffrey M Pina 1 , Janice M Reynaga 1 , Anthony A M Truong 1 , Niroshika M Keppetipola 1
Biochemistry ( IF 2.9 ) Pub Date : 2018-05-31 00:00:00 , DOI: 10.1021/acs.biochem.8b00256
Jeffrey M Pina 1 , Janice M Reynaga 1 , Anthony A M Truong 1 , Niroshika M Keppetipola 1
Affiliation
|
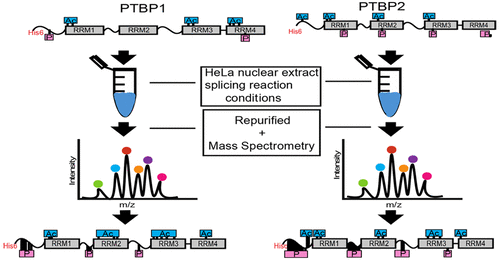
RNA binding proteins play an important role in regulating alternative pre-mRNA splicing and in turn cellular gene expression. Many of these RNA binding proteins occur as gene families with members sharing a high degree of primary structure identity and domain organization yet have tissue-specific expression patterns and regulate different sets of target exons. How highly similar members in a gene family can exert different splicing outcomes is not well understood. We conducted mass spectrometry analysis of post-translational phosphorylation and acetylation modifications for two paralogs of the polypyrimidine tract binding protein family, PTBP1 and PTBP2, to discover modifications that occur in splicing reaction mixtures and to identify discrete modifications that may direct their different splicing activities. We find that PTBP1 and PTBP2 have many distinct phosphate modifications located in the unstructured N-terminal, linker 1, and linker 2 regions. We find that the two proteins have many overlapping acetate modifications in the RNA recognition motifs (RRMs) with a few distinct sites in PTBP1 RRM2 and RRM3. Our data also reveal that lysine residues in the nuclear localization sequence of PTBP2 are acetylated. Collectively, our results highlight important differences in post-translational modifications between the paralogs and suggest a role for them in the differential splicing activity of PTBP1 and PTBP2.
中文翻译:

聚嘧啶束结合蛋白 PTBP1 和 PTBP2 的翻译后修饰
RNA 结合蛋白在调节选择性前 mRNA 剪接进而调节细胞基因表达中起重要作用。这些 RNA 结合蛋白中的许多以基因家族的形式出现,其成员具有高度的一级结构、同一性和结构域组织,但具有组织特异性表达模式并调节不同的靶外显子集。基因家族中高度相似的成员如何产生不同的剪接结果尚不清楚。我们对聚嘧啶束结合蛋白家族的两个旁系同源物 PTBP1 和 PTBP2 的翻译后磷酸化和乙酰化修饰进行了质谱分析,以发现剪接反应混合物中发生的修饰,并鉴定可能指导其不同剪接活性的离散修饰。我们发现 PTBP1 和 PTBP2 在非结构化 N 末端、接头 1 和接头 2 区域具有许多不同的磷酸盐修饰。我们发现这两种蛋白质在 RNA 识别基序 (RRM) 中具有许多重叠的乙酸盐修饰,在 PTBP1、RRM2 和 RRM3 中具有一些不同的位点。我们的数据还显示,PTBP2 核定位序列中的赖氨酸残基被乙酰化。总的来说,我们的结果突出了旁系同源物之间翻译后修饰的重要差异,并表明它们在 PTBP1 和 PTBP2 的差异剪接活性中的作用。
更新日期:2018-05-31
中文翻译:

聚嘧啶束结合蛋白 PTBP1 和 PTBP2 的翻译后修饰
RNA 结合蛋白在调节选择性前 mRNA 剪接进而调节细胞基因表达中起重要作用。这些 RNA 结合蛋白中的许多以基因家族的形式出现,其成员具有高度的一级结构、同一性和结构域组织,但具有组织特异性表达模式并调节不同的靶外显子集。基因家族中高度相似的成员如何产生不同的剪接结果尚不清楚。我们对聚嘧啶束结合蛋白家族的两个旁系同源物 PTBP1 和 PTBP2 的翻译后磷酸化和乙酰化修饰进行了质谱分析,以发现剪接反应混合物中发生的修饰,并鉴定可能指导其不同剪接活性的离散修饰。我们发现 PTBP1 和 PTBP2 在非结构化 N 末端、接头 1 和接头 2 区域具有许多不同的磷酸盐修饰。我们发现这两种蛋白质在 RNA 识别基序 (RRM) 中具有许多重叠的乙酸盐修饰,在 PTBP1、RRM2 和 RRM3 中具有一些不同的位点。我们的数据还显示,PTBP2 核定位序列中的赖氨酸残基被乙酰化。总的来说,我们的结果突出了旁系同源物之间翻译后修饰的重要差异,并表明它们在 PTBP1 和 PTBP2 的差异剪接活性中的作用。


































 京公网安备 11010802027423号
京公网安备 11010802027423号