当前位置:
X-MOL 学术
›
J. Phys. Chem. B
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
用单分子FRET剖析构象动力学调节的酶催化
The Journal of Physical Chemistry B ( IF 2.8 ) Pub Date : 2018-05-31 , DOI: 10.1021/acs.jpcb.8b02374
Shaowen Wu 1 , Jianwei Liu 1 , Wenning Wang 1
The Journal of Physical Chemistry B ( IF 2.8 ) Pub Date : 2018-05-31 , DOI: 10.1021/acs.jpcb.8b02374
Shaowen Wu 1 , Jianwei Liu 1 , Wenning Wang 1
Affiliation
|
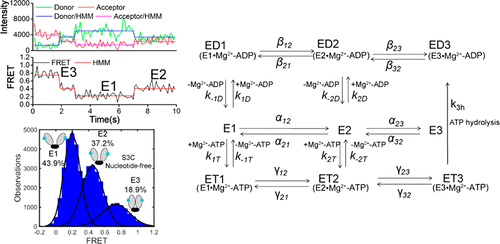
酶蛋白的构象变化通常与催化反应结合并调节酶活性。在这些复杂的情况下,单分子技术是研究酶催化机理的强大工具。然而,在单分子检测实验中不能同时监测化学反应周期和构象变化,从而导致一些未解决的关键动力学参数。在这里,我们描述了一种从综合单分子FRET(smFRET)测量和模型分析中提取所有动力学参数的方法。在smFRET的基础上,我们通过用smFRET测量的构象态总体和状态转变速率常数的输入来求解动力学模型的速率方程,从而计算出无法检测的参数。MalK案例研究2 ATPase证明该方法可以揭示该酶催化反应的定量机理及其偶联的构象动力学。本研究中采用的策略可广泛用于研究其他酶的构象波动耦合催化。

"点击查看英文标题和摘要"
更新日期:2018-06-01

"点击查看英文标题和摘要"



































 京公网安备 11010802027423号
京公网安备 11010802027423号