Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Integrative Analysis of Axolotl Gene Expression Data from Regenerative and Wound Healing Limb Tissues.
Scientific Reports ( IF 3.8 ) Pub Date : 2019-12-30 , DOI: 10.1038/s41598-019-56829-6 Mustafa Sibai 1 , Cüneyd Parlayan 2, 3 , Pelin Tuğlu 2 , Gürkan Öztürk 2, 4 , Turan Demircan 2, 5
Scientific Reports ( IF 3.8 ) Pub Date : 2019-12-30 , DOI: 10.1038/s41598-019-56829-6 Mustafa Sibai 1 , Cüneyd Parlayan 2, 3 , Pelin Tuğlu 2 , Gürkan Öztürk 2, 4 , Turan Demircan 2, 5
Affiliation
|
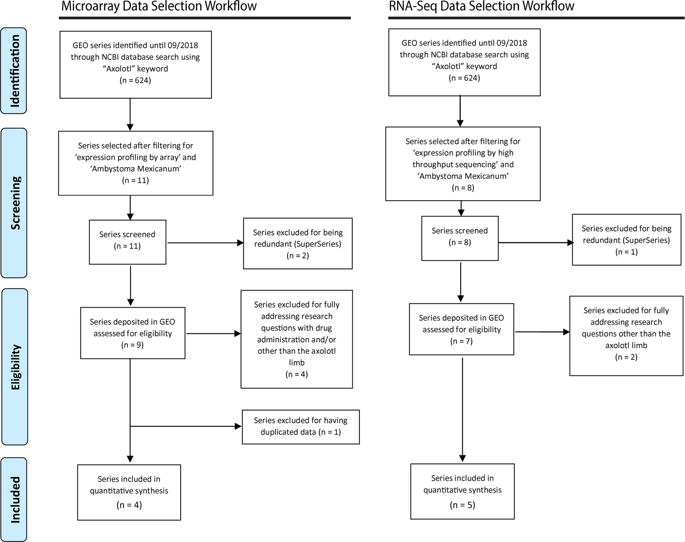
Axolotl (Ambystoma mexicanum) is a urodele amphibian endowed with remarkable regenerative capacities manifested in scarless wound healing and restoration of amputated limbs, which makes it a powerful experimental model for regenerative biology and medicine. Previous studies have utilized microarrays and RNA-Seq technologies for detecting differentially expressed (DE) genes in different phases of the axolotl limb regeneration. However, sufficient consistency may be lacking due to statistical limitations arising from intra-laboratory analyses. This study aims to bridge such gaps by performing an integrative analysis of publicly available microarray and RNA-Seq data from axolotl limb samples having comparable study designs using the "merging" method. A total of 351 genes were found DE in regenerative samples compared to the control in data of both technologies, showing an adjusted p-value < 0.01 and log fold change magnitudes >1. Downstream analyses illustrated consistent correlations of the directionality of DE genes within and between data of both technologies, as well as concordance with the literature on regeneration related biological processes. qRT-PCR analysis validated the observed expression level differences of five of the top DE genes. Future studies may benefit from the utilized concept and approach for enhanced statistical power and robust discovery of biomarkers of regeneration.
中文翻译:

来自再生和伤口愈合肢体组织的蝾螈基因表达数据的综合分析。
蝾螈(Ambystoma mexicanum)是一种有尾目两栖动物,具有卓越的再生能力,表现为无疤痕伤口愈合和截肢恢复,这使其成为再生生物学和医学的强大实验模型。先前的研究利用微阵列和RNA-Seq技术来检测蝾螈肢体再生不同阶段的差异表达(DE)基因。然而,由于实验室内分析产生的统计限制,可能缺乏足够的一致性。本研究旨在通过使用“合并”方法对来自具有可比研究设计的蝾螈肢体样本的公开微阵列和RNA-Seq数据进行综合分析来弥补这些差距。与两种技术的数据中的对照相比,在再生样本中总共发现了 351 个基因 DE,显示调整后的 p 值 < 0.01 和对数倍数变化幅度 >1。下游分析表明两种技术数据内和数据之间 DE 基因方向性的一致相关性,以及与再生相关生物过程的文献的一致性。 qRT-PCR 分析验证了观察到的五个顶级 DE 基因的表达水平差异。未来的研究可能会受益于所使用的概念和方法,以增强统计能力和再生生物标志物的稳健发现。
更新日期:2019-12-30
中文翻译:

来自再生和伤口愈合肢体组织的蝾螈基因表达数据的综合分析。
蝾螈(Ambystoma mexicanum)是一种有尾目两栖动物,具有卓越的再生能力,表现为无疤痕伤口愈合和截肢恢复,这使其成为再生生物学和医学的强大实验模型。先前的研究利用微阵列和RNA-Seq技术来检测蝾螈肢体再生不同阶段的差异表达(DE)基因。然而,由于实验室内分析产生的统计限制,可能缺乏足够的一致性。本研究旨在通过使用“合并”方法对来自具有可比研究设计的蝾螈肢体样本的公开微阵列和RNA-Seq数据进行综合分析来弥补这些差距。与两种技术的数据中的对照相比,在再生样本中总共发现了 351 个基因 DE,显示调整后的 p 值 < 0.01 和对数倍数变化幅度 >1。下游分析表明两种技术数据内和数据之间 DE 基因方向性的一致相关性,以及与再生相关生物过程的文献的一致性。 qRT-PCR 分析验证了观察到的五个顶级 DE 基因的表达水平差异。未来的研究可能会受益于所使用的概念和方法,以增强统计能力和再生生物标志物的稳健发现。






























 京公网安备 11010802027423号
京公网安备 11010802027423号