当前位置:
X-MOL 学术
›
Nat. Mach. Intell.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Prediction of drug combination effects with a minimal set of experiments.
Nature Machine Intelligence ( IF 18.8 ) Pub Date : 2019-12-09 , DOI: 10.1038/s42256-019-0122-4 Aleksandr Ianevski 1, 2 , Anil K Giri 1 , Prson Gautam 1 , Alexander Kononov 1 , Swapnil Potdar 1 , Jani Saarela 1 , Krister Wennerberg 1, 3 , Tero Aittokallio 1, 2, 4
Nature Machine Intelligence ( IF 18.8 ) Pub Date : 2019-12-09 , DOI: 10.1038/s42256-019-0122-4 Aleksandr Ianevski 1, 2 , Anil K Giri 1 , Prson Gautam 1 , Alexander Kononov 1 , Swapnil Potdar 1 , Jani Saarela 1 , Krister Wennerberg 1, 3 , Tero Aittokallio 1, 2, 4
Affiliation
|
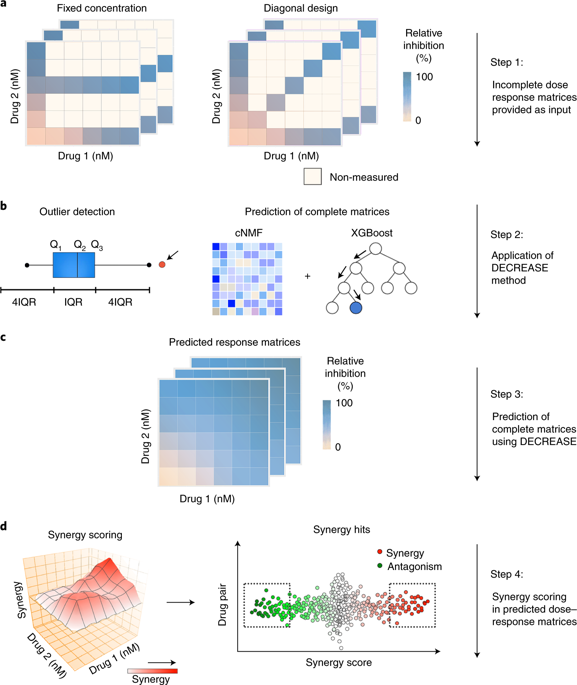
High-throughput drug combination screening provides a systematic strategy to discover unexpected combinatorial synergies in pre-clinical cell models. However, phenotypic combinatorial screening with multi-dose matrix assays is experimentally expensive, especially when the aim is to identify selective combination synergies across a large panel of cell lines or patient samples. Here we implemented DECREASE, an efficient machine learning model that requires only a limited set of pairwise dose-response measurements for accurate prediction of drug combination synergy and antagonism. Using a compendium of 23,595 drug combination matrices tested in various cancer cell lines, and malaria and Ebola infection models, we demonstrate how cost-effective experimental designs with DECREASE capture almost the same degree of information for synergy and antagonism detection as the fully-measured dose-response matrices. Measuring only the diagonal of the matrix provides an accurate and practical option for combinatorial screening. The open-source web-implementation enables applications of DECREASE to both pre-clinical and translational studies.
中文翻译:

用最少的一组实验即可预测药物联合作用。
高通量药物组合筛选为发现临床前细胞模型中意想不到的组合协同作用提供了系统的策略。然而,用多剂量基质测定进行表型组合筛选在实验上是昂贵的,特别是当目的是在一大批细胞系或患者样品中鉴定选择性组合协同作用时。在这里,我们实施了DECREASE,这是一种高效的机器学习模型,仅需要有限的成对剂量响应测量集即可准确预测药物组合的协同作用和拮抗作用。使用在各种癌细胞系以及疟疾和埃博拉病毒感染模型中测试的23,595种药物组合矩阵的纲要,我们展示了使用DECREASE进行的具有成本效益的实验设计如何捕获与完全测量的剂量反应矩阵几乎相同程度的协同和拮抗作用信息。仅测量矩阵的对角线可为组合筛选提供准确而实用的选择。开源Web实施使DECREASE可以应用于临床前研究和翻译研究。
更新日期:2020-01-14
中文翻译:

用最少的一组实验即可预测药物联合作用。
高通量药物组合筛选为发现临床前细胞模型中意想不到的组合协同作用提供了系统的策略。然而,用多剂量基质测定进行表型组合筛选在实验上是昂贵的,特别是当目的是在一大批细胞系或患者样品中鉴定选择性组合协同作用时。在这里,我们实施了DECREASE,这是一种高效的机器学习模型,仅需要有限的成对剂量响应测量集即可准确预测药物组合的协同作用和拮抗作用。使用在各种癌细胞系以及疟疾和埃博拉病毒感染模型中测试的23,595种药物组合矩阵的纲要,我们展示了使用DECREASE进行的具有成本效益的实验设计如何捕获与完全测量的剂量反应矩阵几乎相同程度的协同和拮抗作用信息。仅测量矩阵的对角线可为组合筛选提供准确而实用的选择。开源Web实施使DECREASE可以应用于临床前研究和翻译研究。




















































 京公网安备 11010802027423号
京公网安备 11010802027423号