当前位置:
X-MOL 学术
›
Mol. Phylogenet. Evol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Phylogeny of Diplazium (Athyriaceae) revisited: Resolving the backbone relationships based on plastid genomes and phylogenetic tree space analysis.
Molecular Phylogenetics and Evolution ( IF 3.6 ) Pub Date : 2019-12-04 , DOI: 10.1016/j.ympev.2019.106699 Ran Wei 1 , Xian-Chun Zhang 1
Molecular Phylogenetics and Evolution ( IF 3.6 ) Pub Date : 2019-12-04 , DOI: 10.1016/j.ympev.2019.106699 Ran Wei 1 , Xian-Chun Zhang 1
Affiliation
|
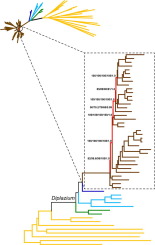
Despite progress in resolving the phylogeny of twinsorus ferns (Diplazium) based on multilocus phylogenetic studies, uncertainty remains especially for deep, or backbone relationships among closely related clades, suggesting a classic case of rapid evolutionary radiation. Here, we investigated the deep phylogenetic relationships within Diplazium by sampling all major clades and using 51 plastid genomes (plastomes), of which 38 were newly sequenced with high-throughput sequencing technology, resulting more than 127,000 informative sites. Using parsimony, maximum likelihood and Bayesian analyses of plastome sequences, we largely resolved the backbone of the phylogeny of Diplazium with strong support. However, we also detected phylogenetic incongruence among different datasets and moderately to poorly supported relationships, particularly at several extremely short internal branches. By using phylogenetic tree space and topology-clustering analyses, we provide evidence that conflicting phylogenetic signals can be found across the trees estimated from individual chloroplast protein-coding genes, which may underlie the difficulty of systematics of Diplazium. Furthermore, our phylogenetic estimate offers more resolution over previous multilocus analyses, providing a framework for future taxonomic revisions of sectional classification of Diplazium. Our study demonstrates the advantage of a character-rich plastome dataset, combining the comparison of different phylogenetic methods, for resolving the recalcitrant lineages that have undergone rapid radiation and dramatic changes in evolutionary rates.
中文翻译:

重新研究系统发育(Athyriaceae):基于质体基因组和系统发育树空间分析解决骨架关系。
尽管基于多基因座系统发育研究在解决双生蕨类植物(双齿蕨)的系统发育方面取得了进展,但是不确定性仍然存在,尤其是对于紧密相关的进化枝之间的深层或骨架关系,这是快速进化辐射的经典案例。在这里,我们通过采样所有主要进化枝并使用51个质体基因组(质体组)来调查Diplazium内的深层系统发育关系,其中38个是通过高通量测序技术新测序的,产生了超过127,000个信息位点。使用简约性,最大似然和塑性组序列的贝叶斯分析,我们在有力的支持下很大程度上分辨了双歧的系统发育主干。但是,我们还检测了不同数据集之间的系统发育不一致,以及中度到不良支持的关系,特别是在几个非常短的内部分支机构。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。我们提供的证据表明,从单个叶绿体蛋白编码基因估计的树木中可以发现有冲突的系统发生信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。我们提供的证据表明,从单个叶绿体蛋白编码基因估计的树木中可以发现有冲突的系统发生信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。为将来对Diplazium的部分分类进行分类修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速率急剧变化的顽固谱系。为将来对Diplazium的部分分类进行分类修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。
更新日期:2019-12-04
中文翻译:

重新研究系统发育(Athyriaceae):基于质体基因组和系统发育树空间分析解决骨架关系。
尽管基于多基因座系统发育研究在解决双生蕨类植物(双齿蕨)的系统发育方面取得了进展,但是不确定性仍然存在,尤其是对于紧密相关的进化枝之间的深层或骨架关系,这是快速进化辐射的经典案例。在这里,我们通过采样所有主要进化枝并使用51个质体基因组(质体组)来调查Diplazium内的深层系统发育关系,其中38个是通过高通量测序技术新测序的,产生了超过127,000个信息位点。使用简约性,最大似然和塑性组序列的贝叶斯分析,我们在有力的支持下很大程度上分辨了双歧的系统发育主干。但是,我们还检测了不同数据集之间的系统发育不一致,以及中度到不良支持的关系,特别是在几个非常短的内部分支机构。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。通过使用系统发育树空间和拓扑聚类分析,我们提供的证据表明,从单个叶绿体蛋白编码基因估计的整个树木中都可以发现相互冲突的系统发育信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。我们提供的证据表明,从单个叶绿体蛋白编码基因估计的树木中可以发现有冲突的系统发生信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。我们提供的证据表明,从单个叶绿体蛋白编码基因估计的树木中可以发现有冲突的系统发生信号,这可能是Diplazium系统学的难点所在。此外,我们的系统发育估计比以前的多基因座分析提供了更高的分辨率,为将来对Diplazium的部分分类进行分类学修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。为将来对Diplazium的部分分类进行分类修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速率急剧变化的顽固谱系。为将来对Diplazium的部分分类进行分类修订提供了框架。我们的研究表明,一个特征丰富的质体数据集的优势,结合了不同系统发育方法的比较,可以解决经历了快速辐射和进化速度急剧变化的顽固谱系。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号