当前位置:
X-MOL 学术
›
Neurobiol. Dis.
›
论文详情
Our official English website, www.x-mol.net, welcomes your feedback! (Note: you will need to create a separate account there.)
元素指纹:重新评估帕金森氏病的脑脊液生物标志物。
Neurobiology of Disease ( IF 5.1 ) Pub Date : 2019-11-13 , DOI: 10.1016/j.nbd.2019.104677 Fabian Maass 1 , Bernhard Michalke 2 , Desiree Willkommen 2 , Andreas Leha 3 , Claudia Schulte 4 , Lars Tönges 5 , Brit Mollenhauer 6 , Claudia Trenkwalder 7 , Daniel Rückamp 8 , Matthias Börger 1 , Inga Zerr 9 , Mathias Bähr 10 , Paul Lingor 11
Neurobiology of Disease ( IF 5.1 ) Pub Date : 2019-11-13 , DOI: 10.1016/j.nbd.2019.104677 Fabian Maass 1 , Bernhard Michalke 2 , Desiree Willkommen 2 , Andreas Leha 3 , Claudia Schulte 4 , Lars Tönges 5 , Brit Mollenhauer 6 , Claudia Trenkwalder 7 , Daniel Rückamp 8 , Matthias Börger 1 , Inga Zerr 9 , Mathias Bähr 10 , Paul Lingor 11
Affiliation
|
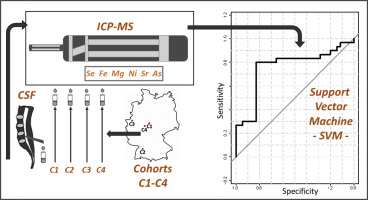
这项研究的目的是基于脑脊髓液(CSF)中的生物元素丰度,验证一种用于分类帕金森氏病(PD)和年龄匹配的对照(AMC)的预测性生物标志物机器学习模型。在这项多中心试验中,参与者来自四个不同的中心。根据标准化协议收集脑脊液。为了确定生物元素,对CSF样品进行了电感耦合等离子体质谱分析。基于六个不同生物元素的水平,将经过先前发现队列训练的预定义支持向量机(SVM)模型应用于区分。包括82名PD患者,68名年龄匹配的对照和7名其他正常压力脑积水(NPH)患者,以验证预定义的SVM模型。六种区分元素(砷,铁,镁,镍,硒,Sr)被定量。基于它们的级别,SVM已成功应用于新的本地队列(AUROC 0.76,灵敏度0.80,特异性0.83),而没有考虑任何其他功能。相同的模型可能没有将PD和AMC / NPH与三个外部队列区分开,这可能是由于中心效应所致。但是,现在可以使用特定于中心的发现队列和交叉验证的方法(具有AUROC 0.78和0.88)对具有完整元素数据集的队列进行区分。合并的PD CSF铁水平与疾病持续时间有明显的相关性(p = .0001)。总之,通过质谱法获得并整合到预测模型中的生物元素CSF模式具有促进PD和AMC分化的潜力。中心偏倚会干扰外部同类群组的应用。

"点击查看英文标题和摘要"
更新日期:2019-11-13

"点击查看英文标题和摘要"






































 京公网安备 11010802027423号
京公网安备 11010802027423号