当前位置:
X-MOL 学术
›
ACS Chem. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Quantitative Analysis of the Substrate Specificity of Human Rhinovirus 3C Protease and Exploration of Its Substrate Recognition Mechanisms.
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2019-10-29 , DOI: 10.1021/acschembio.9b00539 Xian Fan 1 , Xinzhi Li 1 , Yu Zhou 1 , Meng Mei 1 , Pi Liu 2 , Jing Zhao 1 , Wenfang Peng 1 , Zheng-Bing Jiang 1 , Shihui Yang 1 , Brent L Iverson 3 , Guimin Zhang 1 , Li Yi 1
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2019-10-29 , DOI: 10.1021/acschembio.9b00539 Xian Fan 1 , Xinzhi Li 1 , Yu Zhou 1 , Meng Mei 1 , Pi Liu 2 , Jing Zhao 1 , Wenfang Peng 1 , Zheng-Bing Jiang 1 , Shihui Yang 1 , Brent L Iverson 3 , Guimin Zhang 1 , Li Yi 1
Affiliation
|
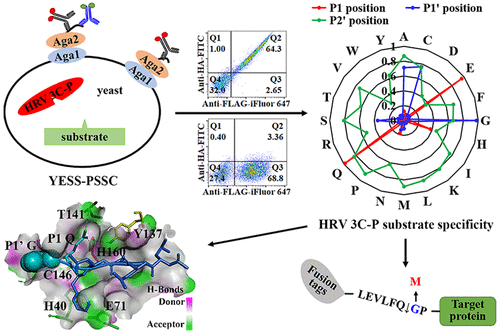
Human rhinovirus 3C protease (HRV 3C-P) is a high-value commercial cysteine protease that could specifically recognize the short peptide sequence of LEVLFQ↓GP. In here, a strategy based on our previous Yeast Endoplasmic Reticulum Sequestration Screening (YESS) approach was developed in Saccharomyces cerevisiae, a model microorganism, to fully characterize the substrate specificity of a typical human virus protease, HRV 3C-P, in a quantitative and fast manner. Our results demonstrated that HRV 3C-P had very high specificity at P1 and P1' positions, only recognizing Gln/Glu at the P1 position and Gly/Ala/Cys/Ser at the P1' position, respectively. Comparably, it exhibited efficient recognition of most residues at the P2' position, except Trp. Further biochemical characterization through site mutagenesis, enzyme structural modeling, and comparison with other 3C proteases indicated that the S1 pocket of HRV 3C-P was constituted by neutral and basic amino acids, in which His160 and Thr141 specifically interacted with Gln or Glu residues at the substrate P1 position. Additionally, the stringent S1' pocket determined its unique property of only accommodating residues without or with short side chains. Based on our characterization, LEVLFQ↓GM was identified as a more favorable substrate than the original LEVLFQ↓GP at high temperature, which might be caused by the conversion of random coils to β-turns in HRV 3C-P along with the temperature increase. Our studies prompted a further understanding of the substrate specificity and recognition mechanism of HRV 3C-P. Besides, the YESS-PSSC combined with the enzyme modeling strategy in this study provides a general strategy for deciphering the substrate specificities of proteases.
中文翻译:

人鼻病毒3C蛋白酶底物特异性的定量分析及其底物识别机制的探索。
人鼻病毒3C蛋白酶(HRV 3C-P)是一种高价值的商业半胱氨酸蛋白酶,可以特异性识别LEVLFQ↓GP的短肽序列。在这里,我们在酿酒酵母(一种模型微生物)中开发了一种基于我们之前的酵母内质网隔离筛选(YESS)方法的策略,以定量和定量的方式充分表征典型人类病毒蛋白酶 HRV 3C-P 的底物特异性。快速的方式。我们的结果表明,HRV 3C-P 在 P1 和 P1' 位点具有非常高的特异性,分别仅识别 P1 位点的 Gln/Glu 和 P1' 位点的 Gly/Ala/Cys/Ser。相比之下,它表现出对 P2' 位除 Trp 之外的大多数残基的有效识别。通过位点突变、酶结构建模以及与其他 3C 蛋白酶的比较进行的进一步生化表征表明,HRV 3C-P 的 S1 口袋由中性和碱性氨基酸构成,其中 His160 和 Thr141 与 Gln 或 Glu 残基特异性相互作用。基板P1位置。此外,严格的S1'口袋决定了其仅容纳不带或带短侧链的残基的独特性质。根据我们的表征,LEVLFQ↓GM 被认为在高温下比原始 LEVLFQ↓GP 更有利,这可能是由于 HRV 3C-P 中的随机线圈随着温度升高而转变为 β 匝所致。我们的研究促使人们进一步了解 HRV 3C-P 的底物特异性和识别机制。此外,本研究中的 YESS-PSSC 与酶建模策略相结合,为破译蛋白酶的底物特异性提供了通用策略。
更新日期:2019-10-29
中文翻译:

人鼻病毒3C蛋白酶底物特异性的定量分析及其底物识别机制的探索。
人鼻病毒3C蛋白酶(HRV 3C-P)是一种高价值的商业半胱氨酸蛋白酶,可以特异性识别LEVLFQ↓GP的短肽序列。在这里,我们在酿酒酵母(一种模型微生物)中开发了一种基于我们之前的酵母内质网隔离筛选(YESS)方法的策略,以定量和定量的方式充分表征典型人类病毒蛋白酶 HRV 3C-P 的底物特异性。快速的方式。我们的结果表明,HRV 3C-P 在 P1 和 P1' 位点具有非常高的特异性,分别仅识别 P1 位点的 Gln/Glu 和 P1' 位点的 Gly/Ala/Cys/Ser。相比之下,它表现出对 P2' 位除 Trp 之外的大多数残基的有效识别。通过位点突变、酶结构建模以及与其他 3C 蛋白酶的比较进行的进一步生化表征表明,HRV 3C-P 的 S1 口袋由中性和碱性氨基酸构成,其中 His160 和 Thr141 与 Gln 或 Glu 残基特异性相互作用。基板P1位置。此外,严格的S1'口袋决定了其仅容纳不带或带短侧链的残基的独特性质。根据我们的表征,LEVLFQ↓GM 被认为在高温下比原始 LEVLFQ↓GP 更有利,这可能是由于 HRV 3C-P 中的随机线圈随着温度升高而转变为 β 匝所致。我们的研究促使人们进一步了解 HRV 3C-P 的底物特异性和识别机制。此外,本研究中的 YESS-PSSC 与酶建模策略相结合,为破译蛋白酶的底物特异性提供了通用策略。

































 京公网安备 11010802027423号
京公网安备 11010802027423号