Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
基于NSGA-II描述符选择的集成hERG数据库的hERG抑制活性支持向量机模型。
Scientific Reports ( IF 3.8 ) Pub Date : 2019-08-21 , DOI: 10.1038/s41598-019-47536-3
Keiji Ogura 1 , Tomohiro Sato 1 , Hitomi Yuki 1 , Teruki Honma 1
Scientific Reports ( IF 3.8 ) Pub Date : 2019-08-21 , DOI: 10.1038/s41598-019-47536-3
Keiji Ogura 1 , Tomohiro Sato 1 , Hitomi Yuki 1 , Teruki Honma 1
Affiliation
|
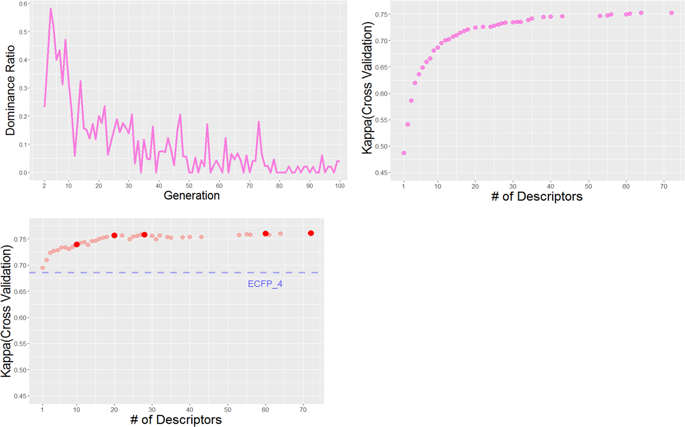
在药物开发计划的早期阶段评估hERG责任很重要。公共数据库中与hERG相关的信息的最新增加使机器学习技术的各种成功应用能够预测hERG抑制。但是,大多数这些研究仅从一个数据库构建数据集,从而限制了模型的可预测性和范围。在这项研究中,使用最大的hERG抑制数据集通过整合多个数据库构建了hERG分类模型。集成的数据集包含来自ChEMBL,GOSTAR,PubChem和hERGCentral的291,000多种结构多样的化合物。预测模型是由支持向量机(SVM)通过基于非支配排序遗传算法-II(NSGA-II)进行描述符选择而建立的,以优化描述符集,从而以最少的描述符数量实现最大的预测性能。使用72个选定的描述符和ECFP_4结构指纹的SVM分类模型记录的测试集的kappa统计值为0.733,准确度为0.984,大大超过了当前商业应用对hERG预测的预测性能。最后,基于训练集和测试集化合物之间的分子相似性,评估了预测模型的适用范围。使用72个选定的描述符和ECFP_4结构指纹的SVM分类模型记录的测试集的kappa统计值为0.733,准确度为0.984,大大超过了当前商业应用对hERG预测的预测性能。最后,基于训练集和测试集化合物之间的分子相似性,评估了预测模型的适用范围。使用72个选定的描述符和ECFP_4结构指纹的SVM分类模型记录的测试集的kappa统计值为0.733,准确度为0.984,大大超过了当前商业应用对hERG预测的预测性能。最后,基于训练集和测试集化合物之间的分子相似性,评估了预测模型的适用范围。

"点击查看英文标题和摘要"
更新日期:2019-08-21

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号