Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
组蛋白-DNA纳米结构在单核苷酸分辨率下基于几何的自组装。
ACS Nano ( IF 15.8 ) Pub Date : 2019-06-20 00:00:00 , DOI: 10.1021/acsnano.9b03259 Maged F Serag,Aimaiti Aikeremu,Ryoko Tsukamoto,Hubert Piwoński,Maram Abadi,Noritada Kaji,Jason R Dwyer,Yoshinobu Baba,Satoshi Habuchi
ACS Nano ( IF 15.8 ) Pub Date : 2019-06-20 00:00:00 , DOI: 10.1021/acsnano.9b03259 Maged F Serag,Aimaiti Aikeremu,Ryoko Tsukamoto,Hubert Piwoński,Maram Abadi,Noritada Kaji,Jason R Dwyer,Yoshinobu Baba,Satoshi Habuchi
|
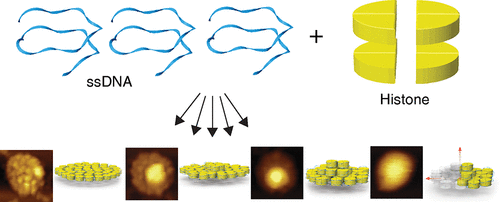
组蛋白是能够与DNA相互作用的基本蛋白质单体,提供了细胞核内DNA压缩的机制。组蛋白和DNA的有序组装过程是构建DNA蛋白质纳米结构的一种潜在候选方法。在这里,利用与序列无关的组蛋白-DNA相互作用,我们提出了一种自组装组蛋白和单链DNA(ssDNA)形成良好定义的组蛋白-DNA(sHD)纳米颗粒及其多维交联复合物(cHD)的方法。 )。通过使用各种分子生物学和显微镜技术,我们阐明了这些复合物的结构,并表明它们是在温度,离子强度,浓度和孵育时间的精心控制条件下形成的。我们还演示了使用一组ssDNA分子标尺和一个几何适应模型,该结果显示sHD和cHD粒子的装配具有精确的几何形状,因此可以通过ssDNA的长度来编程这些粒子中ssDNA的数量。我们进一步表明,cHD的形成会放大ssDNA长度对自组装的影响,从而可以在单个核苷酸分辨率下区分不同长度的ssDNA。我们设想,我们通过几何学指导的自组装组蛋白-DNA纳米结构的方法和基本见识可以作为结构平台,以促进构建精确有序的DNA-蛋白质纳米结构。我们进一步表明,cHD的形成会放大ssDNA长度对自组装的影响,从而可以在单个核苷酸分辨率下区分不同长度的ssDNA。我们设想,我们通过几何学指导的自组装组蛋白-DNA纳米结构的方法和基本见识可以作为结构平台,以促进构建精确有序的DNA-蛋白质纳米结构。我们进一步表明,cHD的形成会放大ssDNA长度对自组装的影响,从而可以在单个核苷酸分辨率下区分不同长度的ssDNA。我们设想,我们通过几何学指导的自组装组蛋白-DNA纳米结构的方法和基本见识可以作为结构平台,以促进构建精确有序的DNA-蛋白质纳米结构。

"点击查看英文标题和摘要"
更新日期:2019-06-20

"点击查看英文标题和摘要"











































 京公网安备 11010802027423号
京公网安备 11010802027423号