当前位置:
X-MOL 学术
›
Biotechnol. J.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Discovery of a Potentially New Subfamily of ELFV Dehydrogenases Effective for l-Arginine Deamination by Enzyme Mining
Biotechnology Journal ( IF 3.2 ) Pub Date : 2017-10-30 , DOI: 10.1002/biot.201700305 Wenjun Wu 1 , Ye Zhang 1 , Jinhai Huang 1 , Yao Wu 1 , Dehua Liu 1, 2 , Zhen Chen 1, 2
Biotechnology Journal ( IF 3.2 ) Pub Date : 2017-10-30 , DOI: 10.1002/biot.201700305 Wenjun Wu 1 , Ye Zhang 1 , Jinhai Huang 1 , Yao Wu 1 , Dehua Liu 1, 2 , Zhen Chen 1, 2
Affiliation
|
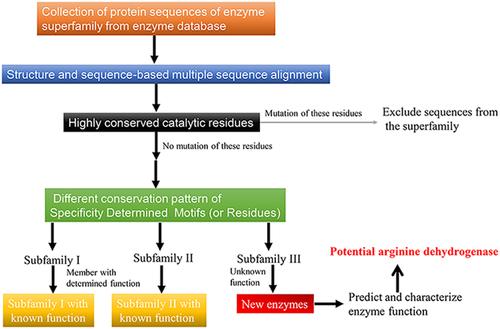
Discovery of enzymes with new functions is very important for de novo pathway design in synthetic biology. Amino acid dehydrogenases catalyze the oxidative deamination of an amino acid to its keto acid, which are widely used for the production of various valuable chemicals. To discover amino acid dehydrogenases with new functions, the authors reevaluate the sequence variability and substrate diversity of ELFV dehydrogenases superfamily in this study. With insights gained from structural and sequential studies, the authors develop an in silico strategy and discover a new category of proteins which are originally annotated as glutamate dehydrogenase but show altered conservation pattern of specificity determined motifs and completely different substrate spectrum. These proteins cannot catalyze the deamination of glutamate and other canonical amino acids except the positively charged amino acid l-arginine, representing a potentially new subfamily of ELFV dehydrogenases. The strategy utilized in this study can also be applied for discovering other useful enzymes. Discovery of enzymes with new functions is very important for de novo pathway design in synthetic biology. In this study, the authors demonstrate that combining structural and sequential alignment and specificity determined motifs for enzyme reannotation and to discover new amino acid dehydrogenases which can effectively catalyze the deamination of arginine. This strategy developed in this work can be used to discover other new enzymes/enzyme functions and reclassification of enzymes.
中文翻译:

发现潜在的新的ELFV脱氢酶亚家族,可通过酶促采矿对1-精氨酸脱氨
发现具有新功能的酶对于合成生物学中的从头途径设计非常重要。氨基酸脱氢酶催化氨基酸氧化为酮酸,广泛用于生产各种有价值的化学物质。为了发现具有新功能的氨基酸脱氢酶,作者在本研究中重新评估了ELFV脱氢酶超家族的序列变异性和底物多样性。利用从结构和顺序研究中获得的见解,作者开发了计算机策略,发现了一类新蛋白质,最初被标注为谷氨酸脱氢酶,但显示出改变的特异性确定的基序和完全不同的底物光谱的保守模式。1-精氨酸,代表潜在的新的ELFV脱氢酶亚家族。本研究中使用的策略也可以用于发现其他有用的酶。发现具有新功能的酶对于合成生物学中的从头途径设计非常重要。在这项研究中,作者证明结合结构和顺序的比对和特异性确定了酶重注释的基序,并发现了可以有效催化精氨酸脱氨的新氨基酸脱氢酶。在这项工作中开发的这种策略可用于发现其他新的酶/酶功能和酶的重新分类。
更新日期:2017-12-14
中文翻译:

发现潜在的新的ELFV脱氢酶亚家族,可通过酶促采矿对1-精氨酸脱氨
发现具有新功能的酶对于合成生物学中的从头途径设计非常重要。氨基酸脱氢酶催化氨基酸氧化为酮酸,广泛用于生产各种有价值的化学物质。为了发现具有新功能的氨基酸脱氢酶,作者在本研究中重新评估了ELFV脱氢酶超家族的序列变异性和底物多样性。利用从结构和顺序研究中获得的见解,作者开发了计算机策略,发现了一类新蛋白质,最初被标注为谷氨酸脱氢酶,但显示出改变的特异性确定的基序和完全不同的底物光谱的保守模式。1-精氨酸,代表潜在的新的ELFV脱氢酶亚家族。本研究中使用的策略也可以用于发现其他有用的酶。发现具有新功能的酶对于合成生物学中的从头途径设计非常重要。在这项研究中,作者证明结合结构和顺序的比对和特异性确定了酶重注释的基序,并发现了可以有效催化精氨酸脱氨的新氨基酸脱氢酶。在这项工作中开发的这种策略可用于发现其他新的酶/酶功能和酶的重新分类。































 京公网安备 11010802027423号
京公网安备 11010802027423号