Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
In Situ Capture of Chromatin Interactions by Biotinylated dCas9.
Cell ( IF 45.5 ) Pub Date : 2017-Aug-24 , DOI: 10.1016/j.cell.2017.08.003 Xin Liu 1 , Yuannyu Zhang 1 , Yong Chen 2 , Mushan Li 3 , Feng Zhou 4 , Kailong Li 1 , Hui Cao 1 , Min Ni 1 , Yuxuan Liu 1 , Zhimin Gu 1 , Kathryn E Dickerson 1 , Shiqi Xie 5 , Gary C Hon 5 , Zhenyu Xuan 2 , Michael Q Zhang 6 , Zhen Shao 3 , Jian Xu 1
Cell ( IF 45.5 ) Pub Date : 2017-Aug-24 , DOI: 10.1016/j.cell.2017.08.003 Xin Liu 1 , Yuannyu Zhang 1 , Yong Chen 2 , Mushan Li 3 , Feng Zhou 4 , Kailong Li 1 , Hui Cao 1 , Min Ni 1 , Yuxuan Liu 1 , Zhimin Gu 1 , Kathryn E Dickerson 1 , Shiqi Xie 5 , Gary C Hon 5 , Zhenyu Xuan 2 , Michael Q Zhang 6 , Zhen Shao 3 , Jian Xu 1
Affiliation
|
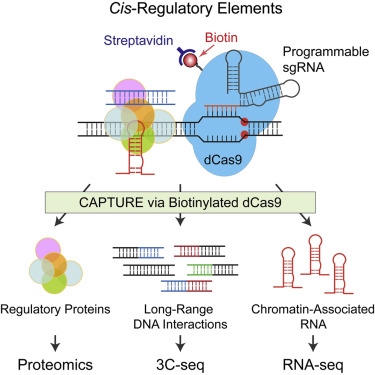
Cis-regulatory elements (CREs) are commonly recognized by correlative chromatin features, yet the molecular composition of the vast majority of CREs in chromatin remains unknown. Here, we describe a CRISPR affinity purification in situ of regulatory elements (CAPTURE) approach to unbiasedly identify locus-specific chromatin-regulating protein complexes and long-range DNA interactions. Using an in vivo biotinylated nuclease-deficient Cas9 protein and sequence-specific guide RNAs, we show high-resolution and selective isolation of chromatin interactions at a single-copy genomic locus. Purification of human telomeres using CAPTURE identifies known and new telomeric factors. In situ capture of individual constituents of the enhancer cluster controlling human β-globin genes establishes evidence for composition-based hierarchical organization. Furthermore, unbiased analysis of chromatin interactions at disease-associated cis-elements and developmentally regulated super-enhancers reveals spatial features that causally control gene transcription. Thus, comprehensive and unbiased analysis of locus-specific regulatory composition provides mechanistic insight into genome structure and function in development and disease.
中文翻译:

通过生物素化 dCas9 原位捕获染色质相互作用。
顺式调控元件 (CRE) 通常通过相关染色质特征来识别,但染色质中绝大多数 CRE 的分子组成仍然未知。在这里,我们描述了一种 CRISPR 亲和纯化原位调节元件 (CAPTURE) 方法,可以公正地识别位点特异性染色质调节蛋白复合物和长程 DNA 相互作用。使用体内生物素化核酸酶缺陷型 Cas9 蛋白和序列特异性引导 RNA,我们展示了单拷贝基因组位点染色质相互作用的高分辨率和选择性分离。使用 CAPTURE 纯化人类端粒可识别已知和新的端粒因子。原位捕获控制人类β-珠蛋白基因的增强子簇的各个成分为基于组成的分层组织奠定了证据。此外,对疾病相关顺式元件和发育调节超级增强子的染色质相互作用的公正分析揭示了因果控制基因转录的空间特征。因此,对位点特异性调控组成的全面且公正的分析提供了对发育和疾病中基因组结构和功能的机制洞察。
更新日期:2017-08-24
中文翻译:

通过生物素化 dCas9 原位捕获染色质相互作用。
顺式调控元件 (CRE) 通常通过相关染色质特征来识别,但染色质中绝大多数 CRE 的分子组成仍然未知。在这里,我们描述了一种 CRISPR 亲和纯化原位调节元件 (CAPTURE) 方法,可以公正地识别位点特异性染色质调节蛋白复合物和长程 DNA 相互作用。使用体内生物素化核酸酶缺陷型 Cas9 蛋白和序列特异性引导 RNA,我们展示了单拷贝基因组位点染色质相互作用的高分辨率和选择性分离。使用 CAPTURE 纯化人类端粒可识别已知和新的端粒因子。原位捕获控制人类β-珠蛋白基因的增强子簇的各个成分为基于组成的分层组织奠定了证据。此外,对疾病相关顺式元件和发育调节超级增强子的染色质相互作用的公正分析揭示了因果控制基因转录的空间特征。因此,对位点特异性调控组成的全面且公正的分析提供了对发育和疾病中基因组结构和功能的机制洞察。































 京公网安备 11010802027423号
京公网安备 11010802027423号