当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
DNA的序列依赖性持久长度
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2017-03-24 00:00:00 , DOI: 10.1021/acs.jctc.6b00904
Jonathan S. Mitchell 1 , Jaroslaw Glowacki 1 , Alexandre E. Grandchamp 1 , Robert S. Manning 2 , John H. Maddocks 1
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2017-03-24 00:00:00 , DOI: 10.1021/acs.jctc.6b00904
Jonathan S. Mitchell 1 , Jaroslaw Glowacki 1 , Alexandre E. Grandchamp 1 , Robert S. Manning 2 , John H. Maddocks 1
Affiliation
|
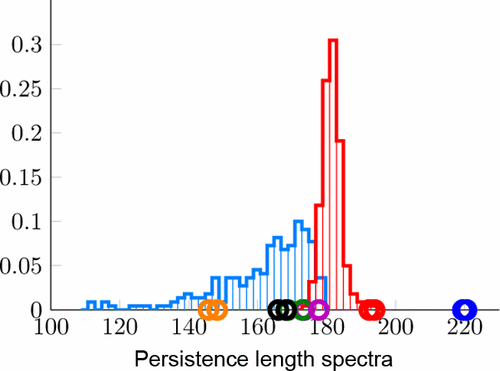
蒙特卡洛代码施加到cgDNA的粗粒硬质基模型B-形式的双链DNA用于预测序列平均持久的长度升˚F在弗洛里的感= 53.5纳米,的升p =在切线-切线相关性衰减的意义上为160 bp或53.5 nm。这些估计值略高于150 bp或50 nm的共识实验值,但鉴于cgDNA,我们认为协议是好的该模型本身是通过分子动力学模拟对长度为10–20 bp的短片段进行参数化的,与持久性长度没有明确的匹配。我们的蒙特卡洛模拟进一步预测,持久性长度可能与 片段的特定序列有很大的依赖关系。我们提出并确认了一个简单的因式分解的数值精度,该因式分解将
片段的特定序列有很大的依赖关系。我们提出并确认了一个简单的因式分解的数值精度,该因式分解将 归因于固有形状的表观正切-正切相关性衰减的部分与
归因于固有形状的表观正切-正切相关性衰减的部分与 纯粹归因于刚度的部分(即所谓的依赖于序列的版本)分开序列平均动态持续长度ļ d(= 58.8的内纳米cgDNA模型)。对于随机和λ噬菌体片段的集合,表观持久性长度的
纯粹归因于刚度的部分(即所谓的依赖于序列的版本)分开序列平均动态持续长度ļ d(= 58.8的内纳米cgDNA模型)。对于随机和λ噬菌体片段的集合,表观持久性长度的 序列标准偏差为4 nm,而我们的动态持久性长度
序列标准偏差为4 nm,而我们的动态持久性长度 的标准偏差仅为1 nm。但是,存在显着的动态余辉长度离群值,包括poly(A)(异常平直和坚硬),poly(TA)(紧密盘绕且异常软)和A相序相序(异常弯曲和僵硬)。我们的数值模拟结果与分子动力学模拟和各种实验数据(包括微圆环化率和立体低温电子显微镜图像)都相当吻合。
的标准偏差仅为1 nm。但是,存在显着的动态余辉长度离群值,包括poly(A)(异常平直和坚硬),poly(TA)(紧密盘绕且异常软)和A相序相序(异常弯曲和僵硬)。我们的数值模拟结果与分子动力学模拟和各种实验数据(包括微圆环化率和立体低温电子显微镜图像)都相当吻合。

"点击查看英文标题和摘要"
更新日期:2017-03-24
 片段的特定序列有很大的依赖关系。我们提出并确认了一个简单的因式分解的数值精度,该因式分解将
片段的特定序列有很大的依赖关系。我们提出并确认了一个简单的因式分解的数值精度,该因式分解将 归因于固有形状的表观正切-正切相关性衰减的部分与
归因于固有形状的表观正切-正切相关性衰减的部分与 纯粹归因于刚度的部分(即所谓的依赖于序列的版本)分开序列平均动态持续长度ļ d(= 58.8的内纳米cgDNA模型)。对于随机和λ噬菌体片段的集合,表观持久性长度的
纯粹归因于刚度的部分(即所谓的依赖于序列的版本)分开序列平均动态持续长度ļ d(= 58.8的内纳米cgDNA模型)。对于随机和λ噬菌体片段的集合,表观持久性长度的 序列标准偏差为4 nm,而我们的动态持久性长度
序列标准偏差为4 nm,而我们的动态持久性长度 的标准偏差仅为1 nm。但是,存在显着的动态余辉长度离群值,包括poly(A)(异常平直和坚硬),poly(TA)(紧密盘绕且异常软)和A相序相序(异常弯曲和僵硬)。我们的数值模拟结果与分子动力学模拟和各种实验数据(包括微圆环化率和立体低温电子显微镜图像)都相当吻合。
的标准偏差仅为1 nm。但是,存在显着的动态余辉长度离群值,包括poly(A)(异常平直和坚硬),poly(TA)(紧密盘绕且异常软)和A相序相序(异常弯曲和僵硬)。我们的数值模拟结果与分子动力学模拟和各种实验数据(包括微圆环化率和立体低温电子显微镜图像)都相当吻合。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号