当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Joining Natural and Synthetic DNA Using Biversal Nucleotides: Efficient Sequencing of Six-Nucleotide DNA
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2024-12-03 , DOI: 10.1021/jacs.4c11043 Bang Wang, Hyo-Joong Kim, Kevin M. Bradley, Cen Chen, Chris McLendon, Zunyi Yang, Steven A. Benner
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2024-12-03 , DOI: 10.1021/jacs.4c11043 Bang Wang, Hyo-Joong Kim, Kevin M. Bradley, Cen Chen, Chris McLendon, Zunyi Yang, Steven A. Benner
|
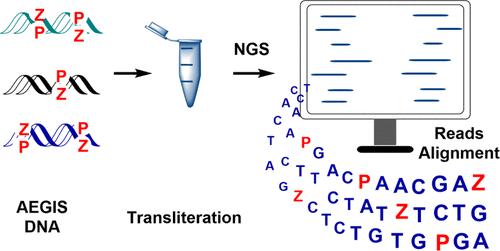
By rearranging hydrogen bond donor and acceptor groups within a standard Watson–Crick geometry, DNA can add eight independently replicable nucleotides forming four additional not found in standard Terran DNA. For many applications, the orthogonal pairing of standard and nonstandard pairs offers a key advantage. However, other applications require standard and nonstandard nucleotides to communicate with each other. This is especially true when seeking to recruit high-throughput instruments (e.g., Illumina), designed to sequence standard 4-nucleotide DNA, to sequence DNA that includes added nucleotides. For this purpose, PCR workflows are needed to replace nonstandard nucleotides in (for example) a 6-letter DNA sequence by defined mixtures of standard nucleotides built from 4 nucleotides. High-throughput sequencing can then report the sequences of those mixtures to bioinformatic alignment tools, which infer the original 6-nucleotide sequence by analysis of the mixtures. Unfortunately, the intrinsic orthogonality of standard and nonstandard nucleotides often demand polymerases that violate pairing biophysics to do this replacement, leading to inefficiencies in this “transliteration” process. Thus, laboratory in vitro evolution (LIVE) using “anthropogenic evolvable genetic information systems” (AEGIS), an important “consumer” of new sequencing tools, has been slow to be democratized; robust sequencing is needed to identify the AegisBodies and AegisZymes that AEGIS-LIVE delivers. This work introduces a new way to connect synthetic and standard molecular biology: biversal nucleotides. In an example presented here, a pyrimidine analogue (pyridine-2-one, y) pairs with Watson–Crick geometry to both a nonstandard base (2-amino-8-imidazo-[1,2a]-1,3,5-triazin-[8H]-4-one, P, the Watson–Crick partner of 6-amino-5-nitro-[1H]-pyridin-2-one, Z) and a base that completes the Watson–Crick hydrogen bond pattern (2-amino-2′-deoxyadenosine, amA). PCR amplification of GACTZP DNA with dyTP delivers products where Z:P pairs are cleanly transliterated to A:T pairs. In parallel, PCR of the same GACTZP sample at higher pH delivers products where Z:P pairs are cleanly transliterated to C:G pairs. By allowing robust sequencing of 6-letter GACTZP DNA, this workflow will help democratize AEGIS-LIVE. Further, other implementations of the biversal concept can enable communication across and between standard DNA and synthetic DNA more generally.
中文翻译:

使用双向核苷酸连接天然 DNA 和合成 DNA:六核苷酸 DNA 的高效测序
通过在标准 Watson-Crick 几何形状中重新排列氢键供体和受体基团,DNA 可以添加 8 个独立可复制的核苷酸,形成 4 个在标准 Terran DNA 中未发现的额外核苷酸。对于许多应用,标准和非标准对的正交配对提供了一个关键优势。然而,其他应用需要标准和非标准核苷酸才能相互通信。当寻求招募旨在对标准 4 核苷酸 DNA 进行测序的高通量仪器(例如 Illumina)来对包含添加核苷酸的 DNA 进行测序时,尤其如此。为此,需要 PCR 工作流程用由 4 个核苷酸构建的确定的标准核苷酸混合物替换(例如)6 个字母的 DNA 序列中的非标准核苷酸。然后,高通量测序可以将这些混合物的序列报告给生物信息学比对工具,这些工具通过分析混合物来推断原始的 6 核苷酸序列。不幸的是,标准核苷酸和非标准核苷酸的内在正交性通常需要违反配对生物物理学的聚合酶来进行这种替换,从而导致这种“音译”过程效率低下。因此,使用“人为进化遗传信息系统”(AEGIS) 的实验室体外进化 (LIVE) 是新测序工具的重要“消费者”,其民主化速度很慢;需要强大的测序来识别 AEGIS-LIVE 提供的 AegisBodies 和 AegisZymes。这项工作引入了一种连接合成和标准分子生物学的新方法:双域核苷酸。 在此处提供的示例中,嘧啶类似物(吡啶-2-酮,y)与 Watson-Crick 几何形状配对到非标准碱基(2-氨基-8-咪唑-[1,2a]-1,3,5-三嗪-[8H]-4-酮,P,6-氨基-5-硝基-[1H]-吡啶-2-酮,Z)的 Watson-Crick 伴侣)和完成 Watson-Crick 氢键模式的碱基(2-氨基-2′-脱氧腺苷, amA) 的使用 dyTP 对 GACTZP DNA 进行 PCR 扩增,可得到 Z:P 对净音译为 A:T 对的产物。同时,在较高 pH 值下对相同的 GACTZP 样品进行 PCR 时,可得到 Z:P 对净音译为 C:G 对的产品。通过允许对 6 个字母的 GACTZP DNA 进行稳健测序,该工作流程将有助于 AEGIS-LIVE 的大众化。此外,双向概念的其他实现可以更普遍地实现标准 DNA 和合成 DNA 之间的通信。
更新日期:2024-12-03
中文翻译:

使用双向核苷酸连接天然 DNA 和合成 DNA:六核苷酸 DNA 的高效测序
通过在标准 Watson-Crick 几何形状中重新排列氢键供体和受体基团,DNA 可以添加 8 个独立可复制的核苷酸,形成 4 个在标准 Terran DNA 中未发现的额外核苷酸。对于许多应用,标准和非标准对的正交配对提供了一个关键优势。然而,其他应用需要标准和非标准核苷酸才能相互通信。当寻求招募旨在对标准 4 核苷酸 DNA 进行测序的高通量仪器(例如 Illumina)来对包含添加核苷酸的 DNA 进行测序时,尤其如此。为此,需要 PCR 工作流程用由 4 个核苷酸构建的确定的标准核苷酸混合物替换(例如)6 个字母的 DNA 序列中的非标准核苷酸。然后,高通量测序可以将这些混合物的序列报告给生物信息学比对工具,这些工具通过分析混合物来推断原始的 6 核苷酸序列。不幸的是,标准核苷酸和非标准核苷酸的内在正交性通常需要违反配对生物物理学的聚合酶来进行这种替换,从而导致这种“音译”过程效率低下。因此,使用“人为进化遗传信息系统”(AEGIS) 的实验室体外进化 (LIVE) 是新测序工具的重要“消费者”,其民主化速度很慢;需要强大的测序来识别 AEGIS-LIVE 提供的 AegisBodies 和 AegisZymes。这项工作引入了一种连接合成和标准分子生物学的新方法:双域核苷酸。 在此处提供的示例中,嘧啶类似物(吡啶-2-酮,y)与 Watson-Crick 几何形状配对到非标准碱基(2-氨基-8-咪唑-[1,2a]-1,3,5-三嗪-[8H]-4-酮,P,6-氨基-5-硝基-[1H]-吡啶-2-酮,Z)的 Watson-Crick 伴侣)和完成 Watson-Crick 氢键模式的碱基(2-氨基-2′-脱氧腺苷, amA) 的使用 dyTP 对 GACTZP DNA 进行 PCR 扩增,可得到 Z:P 对净音译为 A:T 对的产物。同时,在较高 pH 值下对相同的 GACTZP 样品进行 PCR 时,可得到 Z:P 对净音译为 C:G 对的产品。通过允许对 6 个字母的 GACTZP DNA 进行稳健测序,该工作流程将有助于 AEGIS-LIVE 的大众化。此外,双向概念的其他实现可以更普遍地实现标准 DNA 和合成 DNA 之间的通信。




















































 京公网安备 11010802027423号
京公网安备 11010802027423号