当前位置:
X-MOL 学术
›
Anal. Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
PyINETA: Open-Source Platform for INADEQUATE-JRES Integration in NMR Metabolomics
Analytical Chemistry ( IF 6.7 ) Pub Date : 2024-11-19 , DOI: 10.1021/acs.analchem.4c03966 Rahil Taujale, Mario Uchimiya, Chaevien S. Clendinen, Ricardo M. Borges, Christoph W. Turck, Arthur S. Edison
Analytical Chemistry ( IF 6.7 ) Pub Date : 2024-11-19 , DOI: 10.1021/acs.analchem.4c03966 Rahil Taujale, Mario Uchimiya, Chaevien S. Clendinen, Ricardo M. Borges, Christoph W. Turck, Arthur S. Edison
|
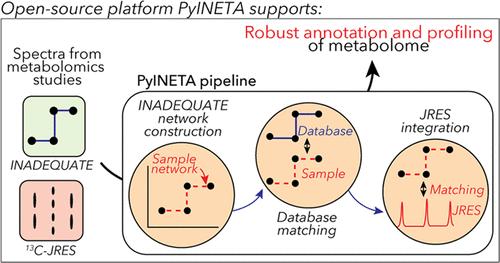
Robust annotation of compounds is a critical element in metabolomics. The 13C-detection NMR experiment incredible natural abundance double-quantum transfer experiment (INADEQUATE) stands out as a powerful tool for structural elucidation, but this valuable experiment is not often included in metabolomics studies. This is partly due to the lack of a community platform that provides structural information based on INADEQUATE. Also, it is often the case that a single study uses various NMR experiments synergistically to improve the quality of information or balance total NMR experiment time, but there is no public platform that can integrate the outputs of INADEQUATE with other NMR experiments. Here, we introduce PyINETA, a Python-based INADEQUATE network analysis. PyINETA is an open-source platform that provides structural information on molecules using INADEQUATE, conducts database searches using an INADEQUATE library, and integrates information on INADEQUATE and a complementary NMR experiment 13C J-resolved experiment (13C-JRES). 13C-JRES was chosen because of its ability to efficiently provide relative quantification in a study of the 13C-enriched samples. Those steps are carried out automatically, and PyINETA keeps track of all the pipeline parameters and outputs, ensuring the transparency of annotation in metabolomics. Our evaluation of PyINETA using a model mouse study showed that PyINETA successfully integrated INADEQUATE and 13C-JRES. The results showed that 13C-labeled amino acids that were fed to mice were transferred to different tissues and were transformed to other metabolites. The distribution of those compounds was tissue-specific, showing enrichment of specific metabolites in the liver, spleen, pancreas, muscle, or lung. PyINETA is freely available on NMRbox.
中文翻译:

PyINETA:用于 NMR 代谢组学中 INADEQUATE-JRES 集成的开源平台
化合物的稳健注释是代谢组学中的一个关键要素。13个 C 检测 NMR 实验令人难以置信的自然丰度双量子转移实验 (INADEQUATE) 作为结构解析的有力工具脱颖而出,但这一有价值的实验通常不包括在代谢组学研究中。这部分是由于缺乏一个基于 INADEQUATE 提供结构信息的社区平台。此外,通常情况下,一项研究协同使用各种 NMR 实验来提高信息质量或平衡 NMR 实验总时间,但没有公共平台可以将 INADEQUATE 的产出与其他 NMR 实验相结合。在这里,我们介绍了 PyINETA,一种基于 Python 的 INADEQUATE 网络分析。PyINETA 是一个开源平台,它使用 INADEQUATE 提供分子的结构信息,使用 INADEQUATE 库进行数据库搜索,并集成有关 INADEQUATE 的信息和互补的 NMR 实验 13C J 分辨实验 (13C-JRES)。13选择 C-JRES 是因为它能够在 13个富含 C 的样品的研究中有效地提供相对定量。这些步骤是自动执行的,PyINETA 会跟踪所有管道参数和输出,确保代谢组学中注释的透明度。我们使用模型小鼠研究对 PyINETA 的评估表明,PyINETA 成功整合了 INADEQUATE 和 13个 C-JRES。结果显示,喂食小鼠的 13个 C 标记氨基酸被转移到不同的组织中,并转化为其他代谢物。 这些化合物的分布是组织特异性的,显示肝脏、脾脏、胰腺、肌肉或肺中特定代谢物的富集。PyINETA 在 NMRbox 上免费提供。
更新日期:2024-11-20
中文翻译:

PyINETA:用于 NMR 代谢组学中 INADEQUATE-JRES 集成的开源平台
化合物的稳健注释是代谢组学中的一个关键要素。13个 C 检测 NMR 实验令人难以置信的自然丰度双量子转移实验 (INADEQUATE) 作为结构解析的有力工具脱颖而出,但这一有价值的实验通常不包括在代谢组学研究中。这部分是由于缺乏一个基于 INADEQUATE 提供结构信息的社区平台。此外,通常情况下,一项研究协同使用各种 NMR 实验来提高信息质量或平衡 NMR 实验总时间,但没有公共平台可以将 INADEQUATE 的产出与其他 NMR 实验相结合。在这里,我们介绍了 PyINETA,一种基于 Python 的 INADEQUATE 网络分析。PyINETA 是一个开源平台,它使用 INADEQUATE 提供分子的结构信息,使用 INADEQUATE 库进行数据库搜索,并集成有关 INADEQUATE 的信息和互补的 NMR 实验 13C J 分辨实验 (13C-JRES)。13选择 C-JRES 是因为它能够在 13个富含 C 的样品的研究中有效地提供相对定量。这些步骤是自动执行的,PyINETA 会跟踪所有管道参数和输出,确保代谢组学中注释的透明度。我们使用模型小鼠研究对 PyINETA 的评估表明,PyINETA 成功整合了 INADEQUATE 和 13个 C-JRES。结果显示,喂食小鼠的 13个 C 标记氨基酸被转移到不同的组织中,并转化为其他代谢物。 这些化合物的分布是组织特异性的,显示肝脏、脾脏、胰腺、肌肉或肺中特定代谢物的富集。PyINETA 在 NMRbox 上免费提供。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号