当前位置:
X-MOL 学术
›
J. Adv. Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Generalized Deep Learning for Histopathology Image Classification Using Supervised Contrastive Learning
Journal of Advanced Research ( IF 11.4 ) Pub Date : 2024-11-16 , DOI: 10.1016/j.jare.2024.11.013 Md Mamunur Rahaman, Ewan K.A. Millar, Erik Meijering
中文翻译:

使用监督对比学习进行组织病理学图像分类的广义深度学习
癌症是世界范围内导致死亡的主要原因,因此需要有效的诊断工具进行早期检测和治疗。组织病理学图像分析对于癌症诊断至关重要,但经常受到人为错误和可变性的阻碍。本研究介绍了 HistopathAI,这是一种专为组织病理学图像分类而设计的混合网络,旨在提高临床病理学的诊断精度和效率。
本研究的主要目标是证明 HistopathAI 利用监督对比学习 (SCL) 和混合深度特征融合 (HDFF),可以显著提高组织病理学图像分类的准确性,包括涉及不平衡数据集的情况。
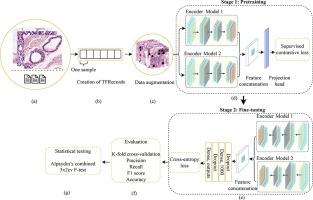
HistopathAI 集成了 EfficientNetB3 和 ResNet50 的功能,使用 HDFF 提供丰富的组织病理学图像表示。该框架采用顺序方法,从特征学习过渡到分类器学习,反映了对比学习的本质,旨在产生卓越的特征表示。该模型将用于特征表示的 SCL 与用于分类的交叉熵 (CE) 损失相结合。我们在 7 个公开可用的数据集和 1 个私有数据集中评估了 HistopathAI,涵盖各个组织病理学领域。
HistopathAI 在所有数据集中实现了最先进的分类准确性,在二进制和多类分类任务中都表现出卓越的性能。统计测试证实,HistopathAI 的性能明显优于基线模型,确保了稳健可靠的改进。
HistopathAI 为组织病理学图像分类提供了强大的工具,提高了诊断准确性并支持向数字病理学的过渡。该框架有可能改善癌症诊断和患者预后,为更广泛的临床应用铺平道路。代码可从 GitHubfootnotehttps://github.com/placeholder-link..
更新日期:2024-11-16
Journal of Advanced Research ( IF 11.4 ) Pub Date : 2024-11-16 , DOI: 10.1016/j.jare.2024.11.013 Md Mamunur Rahaman, Ewan K.A. Millar, Erik Meijering
|
Introduction:
Cancer is a leading cause of death worldwide, necessitating effective diagnostic tools for early detection and treatment. Histopathological image analysis is crucial for cancer diagnosis but is often hindered by human error and variability. This study introduces HistopathAI, a hybrid network designed for histopathology image classification, aimed at enhancing diagnostic precision and efficiency in clinical pathology.Objectives:
The primary goal of this study is to demonstrate that HistopathAI, leveraging supervised contrastive learning (SCL) and hybrid deep feature fusion (HDFF), can significantly improve the accuracy of histopathological image classification, including scenarios involving imbalanced datasets.Methods:
HistopathAI integrates features from EfficientNetB3 and ResNet50, using HDFF to provide a rich representation of histopathology images. The framework employs a sequential methodology, transitioning from feature learning to classifier learning, mirroring the essence of contrastive learning with the aim of producing superior feature representations. The model combines SCL for feature representation with cross-entropy (CE) loss for classification. We evaluated HistopathAI across seven publicly available datasets and one private dataset, covering various histopathology domains.Results:
HistopathAI achieved state-of-the-art classification accuracy across all datasets, demonstrating superior performance in both binary and multiclass classification tasks. Statistical testing confirmed that HistopathAI’s performance is significantly better than baseline models, ensuring robust and reliable improvements.Conclusion:
HistopathAI offers a robust tool for histopathology image classification, enhancing diagnostic accuracy and supporting the transition to digital pathology. This framework has the potential to improve cancer diagnosis and patient outcomes, paving the way for broader clinical application. The code is available from GitHub footnotehttps://github.com/placeholder-link..中文翻译:

使用监督对比学习进行组织病理学图像分类的广义深度学习
介绍:
癌症是世界范围内导致死亡的主要原因,因此需要有效的诊断工具进行早期检测和治疗。组织病理学图像分析对于癌症诊断至关重要,但经常受到人为错误和可变性的阻碍。本研究介绍了 HistopathAI,这是一种专为组织病理学图像分类而设计的混合网络,旨在提高临床病理学的诊断精度和效率。
目标:
本研究的主要目标是证明 HistopathAI 利用监督对比学习 (SCL) 和混合深度特征融合 (HDFF),可以显著提高组织病理学图像分类的准确性,包括涉及不平衡数据集的情况。
方法:
HistopathAI 集成了 EfficientNetB3 和 ResNet50 的功能,使用 HDFF 提供丰富的组织病理学图像表示。该框架采用顺序方法,从特征学习过渡到分类器学习,反映了对比学习的本质,旨在产生卓越的特征表示。该模型将用于特征表示的 SCL 与用于分类的交叉熵 (CE) 损失相结合。我们在 7 个公开可用的数据集和 1 个私有数据集中评估了 HistopathAI,涵盖各个组织病理学领域。
结果:
HistopathAI 在所有数据集中实现了最先进的分类准确性,在二进制和多类分类任务中都表现出卓越的性能。统计测试证实,HistopathAI 的性能明显优于基线模型,确保了稳健可靠的改进。
结论:
HistopathAI 为组织病理学图像分类提供了强大的工具,提高了诊断准确性并支持向数字病理学的过渡。该框架有可能改善癌症诊断和患者预后,为更广泛的临床应用铺平道路。代码可从 GitHubfootnotehttps://github.com/placeholder-link..


















































 京公网安备 11010802027423号
京公网安备 11010802027423号