当前位置:
X-MOL 学术
›
J. Agric. Food Chem.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Barley Grain Proteome Assessment Using Multi-Environment Trial Data and Machine Learning
Journal of Agricultural and Food Chemistry ( IF 5.7 ) Pub Date : 2024-11-13 , DOI: 10.1021/acs.jafc.4c07017 Maany Ramanan, Harmonie Bettenhausen, Gabriela Grigorean, Christine Diepenbrock, Glen Patrick Fox
Journal of Agricultural and Food Chemistry ( IF 5.7 ) Pub Date : 2024-11-13 , DOI: 10.1021/acs.jafc.4c07017 Maany Ramanan, Harmonie Bettenhausen, Gabriela Grigorean, Christine Diepenbrock, Glen Patrick Fox
|
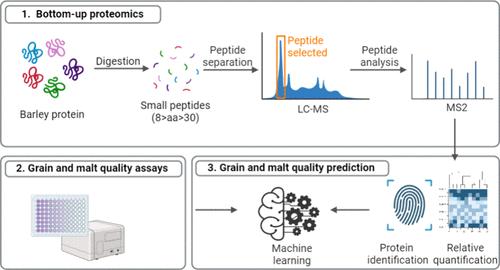
Proteomics can be used to assess individual protein abundances, which could reflect genotypic and environmental effects and potentially predict grain/malt quality. In this study, 79 barley grain samples (genotype-location-year combinations) from Californian multi-environment trials (2017–2022) were assessed using liquid chromatography–mass spectrometry. In total, 3104 proteins were identified across all of the samples. Location, genotype, and year explained 26.7, 17.1, and 14.3% of the variance in the relative abundance of individual proteins, respectively. Sixteen proteins with storage, DNA/RNA binding, or enzymatic functions were significantly higher/lower in abundance (compared to the overall mean) in the Yolo 3 and Imperial Valley locations, Butta 12 and LCS Odyssey genotypes, and the 2017–18 and 2021–22 years. Individual protein abundances were reasonably predictive (RMSECV = 1.25–2.04%) for total, alcohol-soluble, and malt protein content and malt fine extract. This study illustrates the role of the environment in the barley proteome and the utility of proteomics and machine learning to predict grain/malt quality.
中文翻译:

使用多环境试验数据和机器学习进行大麦谷物蛋白质组评估
蛋白质组学可用于评估单个蛋白质丰度,这可以反映基因型和环境影响,并可能预测谷物/麦芽质量。在这项研究中,使用液相色谱-质谱法评估了来自加利福尼亚多环境试验 (2017-2022) 的 79 个大麦谷物样品(基因型-位置-年份组合)。在所有样品中总共鉴定出 3104 种蛋白质。位置、基因型和年份分别解释了单个蛋白质相对丰度的 26.7% 、 17.1% 和 14.3% 的方差。在 Yolo 3 和帝王谷位置、Butta 12 和 LCS Odyssey 基因型以及 2017-18 年和 2021-22 年,16 种具有储存、DNA/RNA 结合或酶促功能的蛋白质的丰度(与总体平均值相比)显着升高/较低。单个蛋白质丰度对总蛋白、醇溶性蛋白和麦芽蛋白含量以及麦芽细提取物具有合理的预测性 (RMSECV = 1.25-2.04%)。本研究说明了环境在大麦蛋白质组中的作用,以及蛋白质组学和机器学习在预测谷物/麦芽质量方面的效用。
更新日期:2024-11-13
中文翻译:

使用多环境试验数据和机器学习进行大麦谷物蛋白质组评估
蛋白质组学可用于评估单个蛋白质丰度,这可以反映基因型和环境影响,并可能预测谷物/麦芽质量。在这项研究中,使用液相色谱-质谱法评估了来自加利福尼亚多环境试验 (2017-2022) 的 79 个大麦谷物样品(基因型-位置-年份组合)。在所有样品中总共鉴定出 3104 种蛋白质。位置、基因型和年份分别解释了单个蛋白质相对丰度的 26.7% 、 17.1% 和 14.3% 的方差。在 Yolo 3 和帝王谷位置、Butta 12 和 LCS Odyssey 基因型以及 2017-18 年和 2021-22 年,16 种具有储存、DNA/RNA 结合或酶促功能的蛋白质的丰度(与总体平均值相比)显着升高/较低。单个蛋白质丰度对总蛋白、醇溶性蛋白和麦芽蛋白含量以及麦芽细提取物具有合理的预测性 (RMSECV = 1.25-2.04%)。本研究说明了环境在大麦蛋白质组中的作用,以及蛋白质组学和机器学习在预测谷物/麦芽质量方面的效用。































 京公网安备 11010802027423号
京公网安备 11010802027423号