当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Recalibration of MARTINI-3 Parameters for Improved Interactions between Peripheral Proteins and Lipid Bilayers
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-11-03 , DOI: 10.1021/acs.jctc.4c00645 Jatin Soni, Shivam Gupta, Taraknath Mandal
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-11-03 , DOI: 10.1021/acs.jctc.4c00645 Jatin Soni, Shivam Gupta, Taraknath Mandal
|
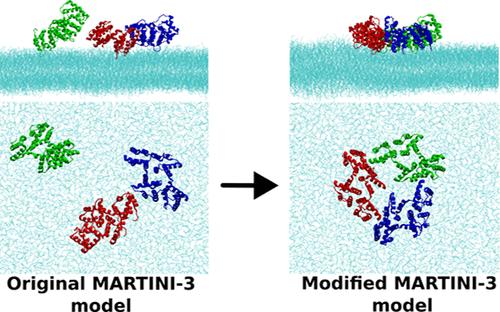
The MARTINI force field is one of the most used coarse-grained models for biomolecular simulations. Many limitations of the model including the protein–protein overaggregation have been improved in its latest version, MARTINI-3. In this study, we investigate the efficacy of the MARTINI-3 parameters for capturing the interactions of peripheral proteins with model plasma membranes. Particularly, we consider two classes of proteins, namely, annexin and epsin, which are known to generate negative and positive membrane curvatures, respectively. We find that current MARTINI-3 parameters are not able to correctly describe the protein–membrane interface and the protein-induced membrane curvatures for any of these proteins. The problem arises due to the lack of proper hydrophobic interactions between the protein residues and lipid tails. Making systematic adjustments, we show that a combination of reduction in the protein–water interactions and enhancement of protein–lipid hydrophobic interactions is essential for accurate prediction of the interfacial structure including the protein-induced membrane curvature. Next, we apply our model to a couple of other peripheral proteins, namely, Snf7, a core component of the ESCRT-III complex, and the PH domain of evectin-2. We find that our model captures the protein–membrane interfacial structure much more accurately than the MARTINI-3 model for all of the peripheral proteins considered in this study. However, the strategy described in this study may not be suitable for oligomeric transmembrane proteins where protein–protein hydrophobic interactions should be increased instead of protein–lipid hydrophobic interactions.
中文翻译:

重新校准 MARTINI-3 参数以改善外周蛋白和脂质双层之间的相互作用
MARTINI 力场是生物分子模拟中最常用的粗粒度模型之一。该模型的许多局限性,包括蛋白质-蛋白质过聚集,在其最新版本 MARTINI-3 中得到了改进。在这项研究中,我们研究了 MARTINI-3 参数捕获外周蛋白与模型质膜相互作用的功效。特别是,我们考虑了两类蛋白质,即 annexin 和 epsin,已知它们分别产生负和正膜曲率。我们发现当前的 MARTINI-3 参数无法正确描述这些蛋白质中的任何一种的蛋白质-膜界面和蛋白质诱导的膜曲率。问题的出现是由于蛋白质残基和脂质尾部之间缺乏适当的疏水相互作用。通过系统调整,我们表明减少蛋白质-水相互作用和增强蛋白质-脂质疏水相互作用的结合对于准确预测界面结构(包括蛋白质诱导的膜曲率)至关重要。接下来,我们将模型应用于其他几种外周蛋白,即 ESCRT-III 复合物的核心成分 Snf7 和 evectin-2 的 PH 结构域。我们发现,对于本研究中考虑的所有外周蛋白,我们的模型比 MARTINI-3 模型更准确地捕获蛋白质-膜界面结构。然而,本研究中描述的策略可能不适用于寡聚跨膜蛋白,其中应增加蛋白质-蛋白质疏水相互作用,而不是蛋白质-脂质疏水相互作用。
更新日期:2024-11-06
中文翻译:

重新校准 MARTINI-3 参数以改善外周蛋白和脂质双层之间的相互作用
MARTINI 力场是生物分子模拟中最常用的粗粒度模型之一。该模型的许多局限性,包括蛋白质-蛋白质过聚集,在其最新版本 MARTINI-3 中得到了改进。在这项研究中,我们研究了 MARTINI-3 参数捕获外周蛋白与模型质膜相互作用的功效。特别是,我们考虑了两类蛋白质,即 annexin 和 epsin,已知它们分别产生负和正膜曲率。我们发现当前的 MARTINI-3 参数无法正确描述这些蛋白质中的任何一种的蛋白质-膜界面和蛋白质诱导的膜曲率。问题的出现是由于蛋白质残基和脂质尾部之间缺乏适当的疏水相互作用。通过系统调整,我们表明减少蛋白质-水相互作用和增强蛋白质-脂质疏水相互作用的结合对于准确预测界面结构(包括蛋白质诱导的膜曲率)至关重要。接下来,我们将模型应用于其他几种外周蛋白,即 ESCRT-III 复合物的核心成分 Snf7 和 evectin-2 的 PH 结构域。我们发现,对于本研究中考虑的所有外周蛋白,我们的模型比 MARTINI-3 模型更准确地捕获蛋白质-膜界面结构。然而,本研究中描述的策略可能不适用于寡聚跨膜蛋白,其中应增加蛋白质-蛋白质疏水相互作用,而不是蛋白质-脂质疏水相互作用。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号