当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
RadicalPy: A Tool for Spin Dynamics Simulations
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-10-29 , DOI: 10.1021/acs.jctc.4c00887 Lewis M. Antill, Emil Vatai
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-10-29 , DOI: 10.1021/acs.jctc.4c00887 Lewis M. Antill, Emil Vatai
|
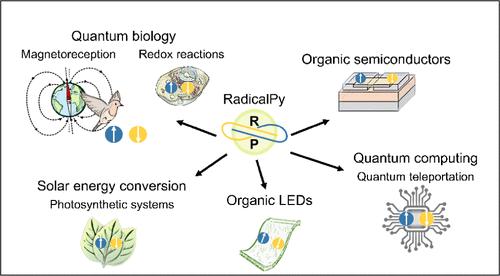
Radical pairs (electron–hole pairs, polaron pairs) are transient reaction intermediates that are found and exploited in all areas of science, from the hard realm of physics in the form of organic semiconductors, spintronics, quantum computing, and solar cells to the soft domain of chemistry and biology under the guise of chemical reactions in solution, biomimetic systems, and quantum biology. Quantitative analysis of radical pair phenomena has historically been successful by a few select groups. With this in mind, we present an intuitive open-source framework in the Python programming language that provides classical, semiclassical, and quantum simulation methodologies. A radical pair kinetic rate equation solver, Monte Carlo-based spin dephasing rate estimations, and molecule database functionalities are implemented. We introduce the kine-quantum method, a new approach that amalgamates classical rate equations, semiclassical, and quantum techniques. This method resolves the prohibitively large memory requirement issues of quantum approaches while achieving higher accuracy, and it also offers wavelength-resolved simulations, producing time- and wavelength-resolved magnetic field effect simulations. Model examples illustrate the versatility and ease of use of the software, including the new approach applied to the magnetosensitive absorption and fluorescence of flavin adenine dinucleotide photochemistry, spin–spin interaction estimation from molecular dynamics simulations on radical pairs inside reverse micelles, radical pair anisotropy inside proteins, and triplet exciton pairs in anthracene crystals. The intuitive interface also allows this software to be used as a teaching or learning aid for those interested in the field of spin chemistry. Furthermore, the software aims to be modular and extensible, with the aim to standardize how spin dynamics simulations are performed.
中文翻译:

RadicalPy:自旋动力学仿真工具
自由基对(电子-空穴对、极化子对)是瞬态反应中间体,在所有科学领域中都有发现和利用,从有机半导体、自旋电子学、量子计算和太阳能电池形式的硬物理学领域,到以溶液中的化学反应为幌子的化学和生物学软领域,仿生系统, 和量子生物学。历史上,少数选定的小组成功地对自由基对现象进行了定量分析。考虑到这一点,我们在 Python 编程语言中提供了一个直观的开源框架,该框架提供经典、半经典和量子模拟方法。实现了自由基对动力学速率方程求解器、基于蒙特卡洛的自旋分离速率估计和分子数据库功能。我们介绍了kine-quantal方法,这是一种融合了经典速率方程、半经典和量子技术的新方法。这种方法解决了量子方法过大的内存需求问题,同时实现了更高的精度,它还提供波长分辨模拟,产生时间和波长分辨的磁场效应模拟。模型示例说明了该软件的多功能性和易用性,包括应用于黄素腺嘌呤二核苷酸光化学磁敏吸收和荧光的新方法、反向胶束内自由基对的分子动力学模拟中的自旋-自旋相互作用估计、蛋白质内部的自由基对各向异性以及蒽晶体中的三重态激子对。直观的界面还允许该软件用作对自旋化学领域感兴趣的人的教学或学习辅助工具。 此外,该软件旨在实现模块化和可扩展性,旨在标准化自旋动力学仿真的执行方式。
更新日期:2024-10-30
中文翻译:

RadicalPy:自旋动力学仿真工具
自由基对(电子-空穴对、极化子对)是瞬态反应中间体,在所有科学领域中都有发现和利用,从有机半导体、自旋电子学、量子计算和太阳能电池形式的硬物理学领域,到以溶液中的化学反应为幌子的化学和生物学软领域,仿生系统, 和量子生物学。历史上,少数选定的小组成功地对自由基对现象进行了定量分析。考虑到这一点,我们在 Python 编程语言中提供了一个直观的开源框架,该框架提供经典、半经典和量子模拟方法。实现了自由基对动力学速率方程求解器、基于蒙特卡洛的自旋分离速率估计和分子数据库功能。我们介绍了kine-quantal方法,这是一种融合了经典速率方程、半经典和量子技术的新方法。这种方法解决了量子方法过大的内存需求问题,同时实现了更高的精度,它还提供波长分辨模拟,产生时间和波长分辨的磁场效应模拟。模型示例说明了该软件的多功能性和易用性,包括应用于黄素腺嘌呤二核苷酸光化学磁敏吸收和荧光的新方法、反向胶束内自由基对的分子动力学模拟中的自旋-自旋相互作用估计、蛋白质内部的自由基对各向异性以及蒽晶体中的三重态激子对。直观的界面还允许该软件用作对自旋化学领域感兴趣的人的教学或学习辅助工具。 此外,该软件旨在实现模块化和可扩展性,旨在标准化自旋动力学仿真的执行方式。




















































 京公网安备 11010802027423号
京公网安备 11010802027423号