当前位置:
X-MOL 学术
›
J. Adv. Res.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
New insights into the enteric methane production based on the archaeal genome atlas of ruminant gastrointestinal tract
Journal of Advanced Research ( IF 11.4 ) Pub Date : 2024-10-18 , DOI: 10.1016/j.jare.2024.09.016 Yifan Tang, Xiaohan Liu, Senlin Zhu, Minghui Jia, Jian-Xin Liu, Hui-Zeng Sun
Journal of Advanced Research ( IF 11.4 ) Pub Date : 2024-10-18 , DOI: 10.1016/j.jare.2024.09.016 Yifan Tang, Xiaohan Liu, Senlin Zhu, Minghui Jia, Jian-Xin Liu, Hui-Zeng Sun
|
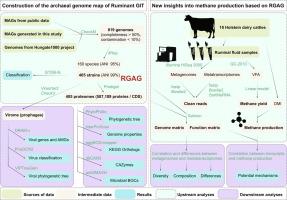
As one of the important components of ruminant gastrointestinal tract (GIT) microbiome, archaea are involved in many biological processes, especially methanogenesis. In spite of being a well-recognised member of the mammalian gut microbiome, it remains poorly characterized, partly due to the lack of a unified reference genome catalog.
中文翻译:

基于反刍动物胃肠道古细菌基因组图谱的肠道甲烷生产新见解
作为反刍动物胃肠道 (GIT) 微生物组的重要组成部分之一,古细菌参与许多生物过程,尤其是甲烷生成。尽管它是哺乳动物肠道微生物组公认的成员,但它的特征仍然很差,部分原因是缺乏统一的参考基因组目录。
更新日期:2024-10-18
中文翻译:

基于反刍动物胃肠道古细菌基因组图谱的肠道甲烷生产新见解
作为反刍动物胃肠道 (GIT) 微生物组的重要组成部分之一,古细菌参与许多生物过程,尤其是甲烷生成。尽管它是哺乳动物肠道微生物组公认的成员,但它的特征仍然很差,部分原因是缺乏统一的参考基因组目录。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号