当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Ramachandran-like Conformational Space for DNA
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-10-18 , DOI: 10.1021/acs.jcim.4c01294 Gabriela da Rosa, Leandro Grille, Pablo D. Dans
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-10-18 , DOI: 10.1021/acs.jcim.4c01294 Gabriela da Rosa, Leandro Grille, Pablo D. Dans
|
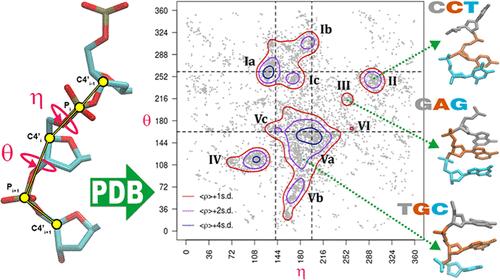
DNA’s ability to exist in a wide variety of structural forms, subforms, and secondary motifs is fundamental to numerous biological processes and has driven the development of biotechnological applications. Major determinants of DNA flexibility are the multiple torsional degrees of freedom around the phosphodiester backbone. This high complexity can be rationalized by using two pseudotorsional angles linking atoms P and C4′, from which Ramachandran-like plots can be built. In this contribution, we explore the distribution of η (eta: C4′i–1-Pi-C4′i-Pi+1) and θ (theta: Pi-C4′i-Pi+1-C4′i+1) angles in known experimental structures retrieved from the Protein Data Bank (PDB), subdividing the conformational space into different datasets. After the removal of the canonical/helical conformations typical of the B-form, we find the existence of a conformational map with clearly permitted and forbidden regions. Some of these regions are populated with specific DNA forms, like Z- or A-DNA, or by specific secondary motifs, like G-quadruplexes and junctions. We evaluated the sequence dependency and energy relationship among the high-density regions identified in the η–θ space. Furthermore, we analyzed the effect produced by proteins and cations when bound to DNA, finding that specific proteins produce some nonhelical conformations, while other regions appear to be stabilized by divalent cations.
中文翻译:

DNA 的拉氏样构象空间
DNA 以多种结构形式、亚形式和次级基序存在的能力是许多生物过程的基础,并推动了生物技术应用的发展。DNA 灵活性的主要决定因素是磷酸二酯骨架周围的多个扭转自由度。这种高复杂性可以通过使用连接原子 P 和 C4' 的两个假扭转角来合理化,从中可以构建类似拉钱德兰的图。在这篇文章中,我们探讨了η (eta: C4′i–1-P-C4′-Pi+1) 和 θ (theta: P-C4′-Pi+1-C4′i+1) 角度在从蛋白质数据库 (PDB) 检索的已知实验结构中的分布,将构象空间细分为不同的数据集。在去除 B 型典型的规范/螺旋构象后,我们发现存在具有明确允许和禁止区域的构象图。其中一些区域填充有特定的 DNA 形式,如 Z 或 A-DNA,或特定的二级基序,如 G 四链体和连接。我们评估了 η-θ 空间中确定的高密度区域之间的序列依赖性和能量关系。此外,我们分析了蛋白质和阳离子与 DNA 结合时产生的效应,发现特定蛋白质产生一些非螺旋构象,而其他区域似乎被二价阳离子稳定。
更新日期:2024-10-18
中文翻译:

DNA 的拉氏样构象空间
DNA 以多种结构形式、亚形式和次级基序存在的能力是许多生物过程的基础,并推动了生物技术应用的发展。DNA 灵活性的主要决定因素是磷酸二酯骨架周围的多个扭转自由度。这种高复杂性可以通过使用连接原子 P 和 C4' 的两个假扭转角来合理化,从中可以构建类似拉钱德兰的图。在这篇文章中,我们探讨了η (eta: C4′i–1-P-C4′-Pi+1) 和 θ (theta: P-C4′-Pi+1-C4′i+1) 角度在从蛋白质数据库 (PDB) 检索的已知实验结构中的分布,将构象空间细分为不同的数据集。在去除 B 型典型的规范/螺旋构象后,我们发现存在具有明确允许和禁止区域的构象图。其中一些区域填充有特定的 DNA 形式,如 Z 或 A-DNA,或特定的二级基序,如 G 四链体和连接。我们评估了 η-θ 空间中确定的高密度区域之间的序列依赖性和能量关系。此外,我们分析了蛋白质和阳离子与 DNA 结合时产生的效应,发现特定蛋白质产生一些非螺旋构象,而其他区域似乎被二价阳离子稳定。


















































 京公网安备 11010802027423号
京公网安备 11010802027423号