当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Toward Reliable Conformational Energies of Amino Acids and Dipeptides─The DipCONFS Benchmark and DipCONL Datasets
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-09-11 , DOI: 10.1021/acs.jctc.4c00801 Christoph Plett 1 , Stefan Grimme 1 , Andreas Hansen 1
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-09-11 , DOI: 10.1021/acs.jctc.4c00801 Christoph Plett 1 , Stefan Grimme 1 , Andreas Hansen 1
Affiliation
|
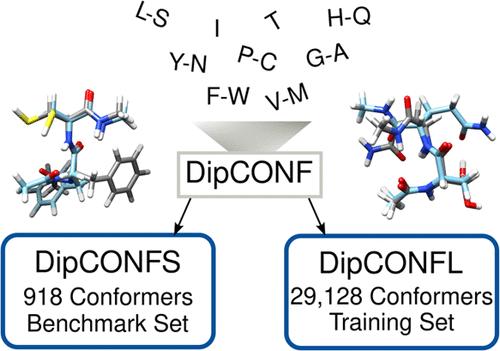
Simulating peptides and proteins is becoming increasingly important, leading to a growing need for efficient computational methods. These are typically semiempirical quantum mechanical (SQM) methods, force fields (FFs), or machine-learned interatomic potentials (MLIPs), all of which require a large amount of accurate data for robust training and evaluation. To assess potential reference methods and complement the available data, we introduce two sets, DipCONFL and DipCONFS, which cover large parts of the conformational space of 17 amino acids and their 289 possible dipeptides in aqueous solution. The conformers were selected from the exhaustive PeptideCS dataset by Andris et al. [ J. Phys. Chem. B 2022, 126, 5949–5958]. The structures, originally generated with GFN2-xTB, were reoptimized using the accurate r2SCAN-3c density functional theory (DFT) composite method including the implicit CPCM water solvation model. The DipCONFS benchmark set contains 918 conformers and is one of the largest sets with highly accurate coupled cluster conformational energies so far. It is employed to evaluate various DFT and wave function theory (WFT) methods, especially regarding whether they are accurate enough to be used as reliable reference methods for larger datasets intended for training and testing more approximated SQM, FF, and MLIP methods. The results reveal that the originally provided BP86-D3(BJ)/DGauss-DZVP conformational energies are not sufficiently accurate. Among the DFT methods tested as an alternative reference level, the revDSD-PBEP86-D4 double hybrid performs best with a mean absolute error (MAD) of 0.2 kcal mol–1 compared with the PNO-LCCSD(T)-F12b reference. The very efficient r2SCAN-3c composite method also shows excellent results, with an MAD of 0.3 kcal mol–1, similar to the best-tested hybrid ωB97M-D4. With these findings, we compiled the large DipCONFL set, which includes over 29,000 realistic conformers in solution with reasonably accurate r2SCAN-3c reference conformational energies, gradients, and further properties potentially relevant for training MLIP methods. This set, also in comparison to DipCONFS, is used to assess the performance of various SQM, FF, and MLIP methods robustly and can complement training sets for those.
中文翻译:

迈向氨基酸和二肽的可靠构象能 — DipCONFS 基准和 DipCONL 数据集
模拟肽和蛋白质变得越来越重要,导致对高效计算方法的需求不断增长。这些通常是半经验量子力学 (SQM) 方法、力场 (FF) 或机器学习原子间势 (MLIP),所有这些都需要大量准确的数据来进行稳健的训练和评估。为了评估潜在的参考方法并补充现有数据,我们引入了两组 DipCONFL 和 DipCONFS,它们覆盖了水溶液中 17 种氨基酸及其 289 种可能的二肽的构象空间的大部分。构象异构体是从 Andris 等人的详尽 PeptideCS 数据集中选择的。 [ J.物理学。化学。 B 2022 , 126 , 5949–5958]。最初使用 GFN2-xTB 生成的结构,使用精确的 r 2 SCAN-3c 密度泛函理论 (DFT) 复合方法(包括隐式 CPCM 水溶剂化模型)重新优化。 DipCONFS 基准集包含 918 个构象异构体,是迄今为止最大的具有高精度耦合簇构象能的集之一。它用于评估各种 DFT 和波函数理论 (WFT) 方法,特别是关于它们是否足够准确,可以用作用于训练和测试更近似的 SQM、FF 和 MLIP 方法的较大数据集的可靠参考方法。结果表明,最初提供的BP86-D3(BJ)/DGauss-DZVP构象能不够准确。在作为替代参考水平进行测试的 DFT 方法中,与 PNO-LCCCD(T)-F12b 参考相比,revDSD-PBEP86-D4 双混合体表现最佳,平均绝对误差 (MAD) 为 0.2 kcal mol –1 。 非常高效的 r 2 SCAN-3c 复合方法也显示出优异的结果,MAD 为 0.3 kcal mol –1 ,类似于经过最佳测试的混合体 ωB97M-D4。根据这些发现,我们编译了大型 DipCONFL 集,其中包括解决方案中的 29,000 多个现实构象异构体,以及相当准确的 r 2 SCAN-3c 参考构象能量、梯度以及与训练 MLIP 方法潜在相关的其他属性。与 DipCONFS 相比,该集用于稳健地评估各种 SQM、FF 和 MLIP 方法的性能,并可以补充这些方法的训练集。
更新日期:2024-09-11
中文翻译:

迈向氨基酸和二肽的可靠构象能 — DipCONFS 基准和 DipCONL 数据集
模拟肽和蛋白质变得越来越重要,导致对高效计算方法的需求不断增长。这些通常是半经验量子力学 (SQM) 方法、力场 (FF) 或机器学习原子间势 (MLIP),所有这些都需要大量准确的数据来进行稳健的训练和评估。为了评估潜在的参考方法并补充现有数据,我们引入了两组 DipCONFL 和 DipCONFS,它们覆盖了水溶液中 17 种氨基酸及其 289 种可能的二肽的构象空间的大部分。构象异构体是从 Andris 等人的详尽 PeptideCS 数据集中选择的。 [ J.物理学。化学。 B 2022 , 126 , 5949–5958]。最初使用 GFN2-xTB 生成的结构,使用精确的 r 2 SCAN-3c 密度泛函理论 (DFT) 复合方法(包括隐式 CPCM 水溶剂化模型)重新优化。 DipCONFS 基准集包含 918 个构象异构体,是迄今为止最大的具有高精度耦合簇构象能的集之一。它用于评估各种 DFT 和波函数理论 (WFT) 方法,特别是关于它们是否足够准确,可以用作用于训练和测试更近似的 SQM、FF 和 MLIP 方法的较大数据集的可靠参考方法。结果表明,最初提供的BP86-D3(BJ)/DGauss-DZVP构象能不够准确。在作为替代参考水平进行测试的 DFT 方法中,与 PNO-LCCCD(T)-F12b 参考相比,revDSD-PBEP86-D4 双混合体表现最佳,平均绝对误差 (MAD) 为 0.2 kcal mol –1 。 非常高效的 r 2 SCAN-3c 复合方法也显示出优异的结果,MAD 为 0.3 kcal mol –1 ,类似于经过最佳测试的混合体 ωB97M-D4。根据这些发现,我们编译了大型 DipCONFL 集,其中包括解决方案中的 29,000 多个现实构象异构体,以及相当准确的 r 2 SCAN-3c 参考构象能量、梯度以及与训练 MLIP 方法潜在相关的其他属性。与 DipCONFS 相比,该集用于稳健地评估各种 SQM、FF 和 MLIP 方法的性能,并可以补充这些方法的训练集。

































 京公网安备 11010802027423号
京公网安备 11010802027423号