当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
IEV2Mol: Molecular Generative Model Considering Protein–Ligand Interaction Energy Vectors
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-09-10 , DOI: 10.1021/acs.jcim.4c00842 Mami Ozawa 1 , Shogo Nakamura 2 , Nobuaki Yasuo 3 , Masakazu Sekijima 1
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-09-10 , DOI: 10.1021/acs.jcim.4c00842 Mami Ozawa 1 , Shogo Nakamura 2 , Nobuaki Yasuo 3 , Masakazu Sekijima 1
Affiliation
|
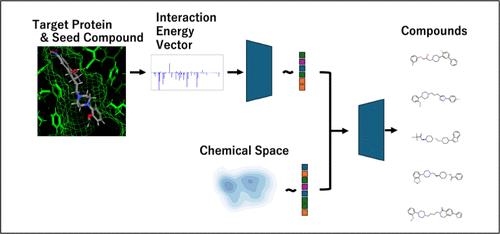
Generating drug candidates with desired protein–ligand interactions is a significant challenge in structure-based drug design. In this study, a new generative model, IEV2Mol, is proposed that incorporates interaction energy vectors (IEVs) between proteins and ligands obtained from docking simulations, which quantitatively capture the strength of each interaction type, such as hydrogen bonds, electrostatic interactions, and van der Waals forces. By integrating this IEV into an end-to-end variational autoencoder (VAE) framework that learns the chemical space from SMILES and minimizes the reconstruction error of the SMILES, the model can more accurately generate compounds with the desired interactions. To evaluate the effectiveness of IEV2Mol, we performed benchmark comparisons with randomly selected compounds, unconstrained VAE models (JT-VAE), and compounds generated by RNN models based on interaction fingerprints (IFP-RNN). The results show that the compounds generated by IEV2Mol retain a significantly greater percentage of the binding mode of the query structure than those of the other methods. Furthermore, IEV2Mol was able to generate compounds with interactions similar to those of the input compounds, regardless of structural similarity. The source code and trained models for IEV2Mol, JT-VAE, and IFP-RNN designed for generating compounds active against the DRD2, AA2AR, and AKT1, as well as the data sets (DM-QP-1M, active compounds to each protein, and ChEMBL33) utilized in this study, are released under the MIT License and available at https://github.com/sekijima-lab/IEV2Mol.
中文翻译:

IEV2Mol:考虑蛋白质-配体相互作用能量载体的分子生成模型
生成具有所需蛋白质-配体相互作用的候选药物是基于结构的药物设计中的重大挑战。在这项研究中,提出了一种新的生成模型 IEV2Mol,该模型结合了通过对接模拟获得的蛋白质和配体之间的相互作用能量向量(IEV),该模型定量捕获每种相互作用类型的强度,例如氢键、静电相互作用和范式相互作用。德瓦尔斯力。通过将此 IEV 集成到端到端变分自动编码器 (VAE) 框架中,该框架从 SMILES 中学习化学空间并最小化 SMILES 的重建误差,该模型可以更准确地生成具有所需相互作用的化合物。为了评估 IEV2Mol 的有效性,我们与随机选择的化合物、无约束 VAE 模型 (JT-VAE) 以及基于相互作用指纹的 RNN 模型生成的化合物 (IFP-RNN) 进行了基准比较。结果表明,与其他方法相比,IEV2Mol 生成的化合物保留了查询结构的结合模式的百分比明显更高。此外,IEV2Mol 能够生成与输入化合物具有相似相互作用的化合物,无论结构是否相似。 IEV2Mol、JT-VAE 和 IFP-RNN 的源代码和训练模型旨在生成针对 DRD2、AA2AR 和 AKT1 的活性化合物,以及数据集(DM-QP-1M、每种蛋白质的活性化合物、本研究中使用的 ChEMBL33 和 ChEMBL33)根据 MIT 许可证发布,可在 https://github.com/sekijima-lab/IEV2Mol 上获取。
更新日期:2024-09-10
中文翻译:

IEV2Mol:考虑蛋白质-配体相互作用能量载体的分子生成模型
生成具有所需蛋白质-配体相互作用的候选药物是基于结构的药物设计中的重大挑战。在这项研究中,提出了一种新的生成模型 IEV2Mol,该模型结合了通过对接模拟获得的蛋白质和配体之间的相互作用能量向量(IEV),该模型定量捕获每种相互作用类型的强度,例如氢键、静电相互作用和范式相互作用。德瓦尔斯力。通过将此 IEV 集成到端到端变分自动编码器 (VAE) 框架中,该框架从 SMILES 中学习化学空间并最小化 SMILES 的重建误差,该模型可以更准确地生成具有所需相互作用的化合物。为了评估 IEV2Mol 的有效性,我们与随机选择的化合物、无约束 VAE 模型 (JT-VAE) 以及基于相互作用指纹的 RNN 模型生成的化合物 (IFP-RNN) 进行了基准比较。结果表明,与其他方法相比,IEV2Mol 生成的化合物保留了查询结构的结合模式的百分比明显更高。此外,IEV2Mol 能够生成与输入化合物具有相似相互作用的化合物,无论结构是否相似。 IEV2Mol、JT-VAE 和 IFP-RNN 的源代码和训练模型旨在生成针对 DRD2、AA2AR 和 AKT1 的活性化合物,以及数据集(DM-QP-1M、每种蛋白质的活性化合物、本研究中使用的 ChEMBL33 和 ChEMBL33)根据 MIT 许可证发布,可在 https://github.com/sekijima-lab/IEV2Mol 上获取。































 京公网安备 11010802027423号
京公网安备 11010802027423号