当前位置:
X-MOL 学术
›
J. Hazard. Mater.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Metagenomic analysis of soil from landfill site reveals a diverse microbial community involved in plastic degradation
Journal of Hazardous Materials ( IF 12.2 ) Pub Date : 2024-09-10 , DOI: 10.1016/j.jhazmat.2024.135804 Akhilesh Kumar , Sudarshan Singh Lakhawat , Kashmir Singh , Vikram Kumar , Kumar Shambhav Verma , Umesh Dwivedi , S.L. Kothari , Naveen Malik , Pushpender Kumar Sharma
Journal of Hazardous Materials ( IF 12.2 ) Pub Date : 2024-09-10 , DOI: 10.1016/j.jhazmat.2024.135804 Akhilesh Kumar , Sudarshan Singh Lakhawat , Kashmir Singh , Vikram Kumar , Kumar Shambhav Verma , Umesh Dwivedi , S.L. Kothari , Naveen Malik , Pushpender Kumar Sharma
|
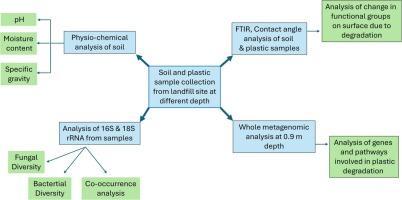
In this study, we have investigated microbial communities structure and function using high throughput amplicon sequencing and whole metagenomic sequencing of DNA extracted from different depths of a plastic-laden landfill site. With diverse taxonomic groups inhabiting the plastic-rich soil, our study demonstrates the remarkable adaptability of microbes to use this new substrate as a carbon source. FTIR spectroscopic analysis of soil indicated degradation of plastic as perceived from the carbonyl index of 0.16, 0.72, and 0.44 at 0.6, 0.9 and 1.2 m depth, respectively. Similarly, water contact angles of 108.7 degree, 99.7 degree, 62.7 degree, and 77.8 degree of plastic pieces collected at 0.3, 0.6, 0.9, and 1.2 m depths respectively showed increased wettability and hydrophilicity of the plastic. Amplicon analysis of 16S and 18 S rRNA revealed a high abundance of several plastic-degrading bacterial groups, including Pseudomonas , Rhizobiales , Micrococcaceae , Chaetomium , Methylocaldum , Micromonosporaceae , Rhodothermaceae and fungi, including Trichoderma , Aspergillus , Candida at 0.9 m. The co-existence of specific microbial groups at different depths of landfill site indicates importance of bacterial and fungal interactions for plastic. Whole metagenome analysis of soil sample at 0.9 m depth revealed a high abundance of genes encoding enzymes that participate in the biodegradation of PVC, polyethylene, PET, and polyurethane. Curation of the pathways related to the degradation of these materials provided a blueprint for plastic biodegradation in this ecosystem. Altogether, our study has highlighted the importance of microbial cooperation for the biodegradation of pollutants. Our metagenome-based investigation supports the current perception that consortia of fungi-bacteria are preferable to axenic cultures for effective bioremediation of the environment.
中文翻译:

对垃圾填埋场土壤的宏基因组分析揭示了参与塑料降解的多种微生物群落
在这项研究中,我们使用高通量扩增子测序和从充满塑料的垃圾填埋场不同深度提取的 DNA 的全宏基因组测序来研究微生物群落的结构和功能。由于不同的分类群栖息在富含塑料的土壤中,我们的研究表明微生物使用这种新基质作为碳源具有非凡的适应性。土壤的 FTIR 光谱分析表明,在 0.6、0.9 和 1.2 m 深度,羰基指数分别为 0.16、0.72 和 0.44,可感知到塑料降解。同样,在 0.3、0.6、0.9 和 1.2 m 深度收集的塑料片的 108.7 度、99.7 度、62.7 度和 77.8 度的水接触角表明塑料的润湿性和亲水性增加。16S 和 18 S rRNA 的扩增子分析显示,几个可塑降解的细菌群丰度很高,包括假单胞菌属、根际菌属、微球菌科、毛壳菌属、甲基球菌属、微单孢子菌科、红磨砂菌科和真菌,包括木霉菌属、曲霉属、念珠菌属 在 0.9 m 处。特定微生物群在垃圾填埋场的不同深度共存表明细菌和真菌相互作用对塑料的重要性。对 0.9 m 深度土壤样本的全宏基因组分析显示,编码参与 PVC、聚乙烯、PET 和聚氨酯生物降解的酶的基因丰度很高。对与这些材料降解相关的途径的整理为该生态系统中的塑料生物降解提供了蓝图。总而言之,我们的研究强调了微生物合作对污染物生物降解的重要性。 我们基于宏基因组的研究支持当前的看法,即真菌细菌群落比轴系培养物更可取,可以对环境进行有效的生物修复。
更新日期:2024-09-10
中文翻译:

对垃圾填埋场土壤的宏基因组分析揭示了参与塑料降解的多种微生物群落
在这项研究中,我们使用高通量扩增子测序和从充满塑料的垃圾填埋场不同深度提取的 DNA 的全宏基因组测序来研究微生物群落的结构和功能。由于不同的分类群栖息在富含塑料的土壤中,我们的研究表明微生物使用这种新基质作为碳源具有非凡的适应性。土壤的 FTIR 光谱分析表明,在 0.6、0.9 和 1.2 m 深度,羰基指数分别为 0.16、0.72 和 0.44,可感知到塑料降解。同样,在 0.3、0.6、0.9 和 1.2 m 深度收集的塑料片的 108.7 度、99.7 度、62.7 度和 77.8 度的水接触角表明塑料的润湿性和亲水性增加。16S 和 18 S rRNA 的扩增子分析显示,几个可塑降解的细菌群丰度很高,包括假单胞菌属、根际菌属、微球菌科、毛壳菌属、甲基球菌属、微单孢子菌科、红磨砂菌科和真菌,包括木霉菌属、曲霉属、念珠菌属 在 0.9 m 处。特定微生物群在垃圾填埋场的不同深度共存表明细菌和真菌相互作用对塑料的重要性。对 0.9 m 深度土壤样本的全宏基因组分析显示,编码参与 PVC、聚乙烯、PET 和聚氨酯生物降解的酶的基因丰度很高。对与这些材料降解相关的途径的整理为该生态系统中的塑料生物降解提供了蓝图。总而言之,我们的研究强调了微生物合作对污染物生物降解的重要性。 我们基于宏基因组的研究支持当前的看法,即真菌细菌群落比轴系培养物更可取,可以对环境进行有效的生物修复。































 京公网安备 11010802027423号
京公网安备 11010802027423号