当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
OLIVES: A Go̅-like Model for Stabilizing Protein Structure via Hydrogen Bonding Native Contacts in the Martini 3 Coarse-Grained Force Field
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-09-05 , DOI: 10.1021/acs.jctc.4c00553 Kasper B. Pedersen 1 , Luís Borges-Araújo 2, 3 , Amanda D. Stange 1 , Paulo C. T. Souza 2, 3 , Siewert J. Marrink 4 , Birgit Schiøtt 1, 5
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-09-05 , DOI: 10.1021/acs.jctc.4c00553 Kasper B. Pedersen 1 , Luís Borges-Araújo 2, 3 , Amanda D. Stange 1 , Paulo C. T. Souza 2, 3 , Siewert J. Marrink 4 , Birgit Schiøtt 1, 5
Affiliation
|
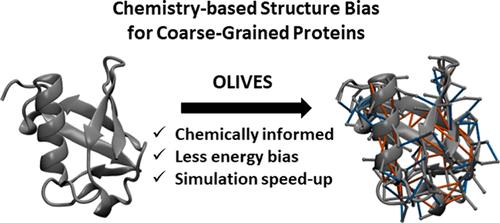
Coarse-grained molecular dynamics simulations enable the modeling of increasingly complex systems at millisecond timescales. The transferable coarse-grained force field Martini 3 has shown great promise in modeling a wide range of biochemical processes, yet folded proteins in Martini 3 are not stable without the application of external bias potentials, such as elastic networks or Go̅-like models. We herein develop an algorithm, called OLIVES, which identifies native contacts with hydrogen bond capabilities in coarse-grained proteins and use it to implement a novel Go̅-like model for Martini 3. We show that the protein structure instability originates in part from the lack of hydrogen bond energy in the coarse-grained force field representation. By using realistic hydrogen bond energies obtained from literature ab initio calculations, it is demonstrated that protein stability can be recovered by the reintroduction of a coarse-grained hydrogen bond network and that OLIVES removes the need for secondary structure restraints. OLIVES is validated against known protein complexes and at the same time addresses the open question of whether there is a need for protein quaternary structure bias in Martini 3 simulations. It is shown that OLIVES can reduce the number of bias terms, hereby speeding up Martini 3 simulations of proteins by up to ≈30% on a GPU architecture compared to the established Go̅MARTINI Go̅-like model.
中文翻译:

OLIVES:通过 Martini 3 粗粒力场中的氢键天然接触稳定蛋白质结构的 Go̅ 类模型
粗粒度分子动力学模拟能够在毫秒时间尺度上对日益复杂的系统进行建模。可转移的粗粒度力场 Martini 3 在模拟各种生化过程方面显示出巨大的前景,但如果不应用外部偏置电位(例如弹性网络或 Go̅ 类模型),Martini 3 中的折叠蛋白质就不稳定。我们在此开发了一种名为 OLIVES 的算法,该算法可识别粗粒蛋白中与氢键功能的天然接触,并使用它为 Martini 3 实现一种新颖的 Go̅ 样模型。我们表明,蛋白质结构的不稳定性部分源于缺乏粗粒力场表示中的氢键能。通过使用从文献从头计算获得的真实氢键能量,证明可以通过重新引入粗粒度氢键网络来恢复蛋白质稳定性,并且 OLIVES 消除了对二级结构限制的需要。 OLIVES 针对已知的蛋白质复合物进行了验证,同时解决了 Martini 3 模拟中是否需要蛋白质四级结构偏差的悬而未决的问题。结果表明,与已建立的 Go̅MARTINI Go̅ 类模型相比,OLIVES 可以减少偏差项的数量,从而在 GPU 架构上将 Martini 3 蛋白质模拟速度提高约 30%。
更新日期:2024-09-05
中文翻译:

OLIVES:通过 Martini 3 粗粒力场中的氢键天然接触稳定蛋白质结构的 Go̅ 类模型
粗粒度分子动力学模拟能够在毫秒时间尺度上对日益复杂的系统进行建模。可转移的粗粒度力场 Martini 3 在模拟各种生化过程方面显示出巨大的前景,但如果不应用外部偏置电位(例如弹性网络或 Go̅ 类模型),Martini 3 中的折叠蛋白质就不稳定。我们在此开发了一种名为 OLIVES 的算法,该算法可识别粗粒蛋白中与氢键功能的天然接触,并使用它为 Martini 3 实现一种新颖的 Go̅ 样模型。我们表明,蛋白质结构的不稳定性部分源于缺乏粗粒力场表示中的氢键能。通过使用从文献从头计算获得的真实氢键能量,证明可以通过重新引入粗粒度氢键网络来恢复蛋白质稳定性,并且 OLIVES 消除了对二级结构限制的需要。 OLIVES 针对已知的蛋白质复合物进行了验证,同时解决了 Martini 3 模拟中是否需要蛋白质四级结构偏差的悬而未决的问题。结果表明,与已建立的 Go̅MARTINI Go̅ 类模型相比,OLIVES 可以减少偏差项的数量,从而在 GPU 架构上将 Martini 3 蛋白质模拟速度提高约 30%。































 京公网安备 11010802027423号
京公网安备 11010802027423号