当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Drug–Target Interactions Prediction at Scale: The Komet Algorithm with the LCIdb Dataset
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-09-05 , DOI: 10.1021/acs.jcim.4c00422 Gwenn Guichaoua 1, 2, 3 , Philippe Pinel 1, 2, 3, 4 , Brice Hoffmann 4 , Chloé-Agathe Azencott 1, 2, 3 , Véronique Stoven 1, 2, 3
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-09-05 , DOI: 10.1021/acs.jcim.4c00422 Gwenn Guichaoua 1, 2, 3 , Philippe Pinel 1, 2, 3, 4 , Brice Hoffmann 4 , Chloé-Agathe Azencott 1, 2, 3 , Véronique Stoven 1, 2, 3
Affiliation
|
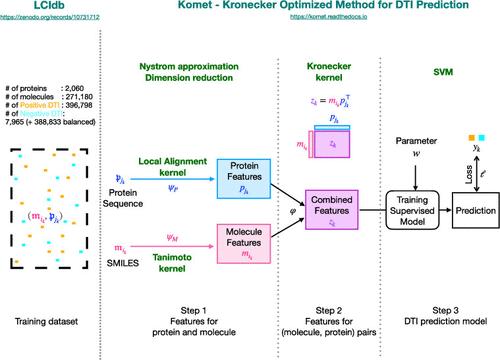
Drug–target interactions (DTIs) prediction algorithms are used at various stages of the drug discovery process. In this context, specific problems such as deorphanization of a new therapeutic target or target identification of a drug candidate arising from phenotypic screens require large-scale predictions across the protein and molecule spaces. DTI prediction heavily relies on supervised learning algorithms that use known DTIs to learn associations between molecule and protein features, allowing for the prediction of new interactions based on learned patterns. The algorithms must be broadly applicable to enable reliable predictions, even in regions of the protein or molecule spaces where data may be scarce. In this paper, we address two key challenges to fulfill these goals: building large, high-quality training datasets and designing prediction methods that can scale, in order to be trained on such large datasets. First, we introduce LCIdb, a curated, large-sized dataset of DTIs, offering extensive coverage of both the molecule and druggable protein spaces. Notably, LCIdb contains a much higher number of molecules than publicly available benchmarks, expanding coverage of the molecule space. Second, we propose Komet (Kronecker Optimized METhod), a DTI prediction pipeline designed for scalability without compromising performance. Komet leverages a three-step framework, incorporating efficient computation choices tailored for large datasets and involving the Nyström approximation. Specifically, Komet employs a Kronecker interaction module for (molecule, protein) pairs, which efficiently captures determinants in DTIs, and whose structure allows for reduced computational complexity and quasi-Newton optimization, ensuring that the model can handle large training sets, without compromising on performance. Our method is implemented in open-source software, leveraging GPU parallel computation for efficiency. We demonstrate the interest of our pipeline on various datasets, showing that Komet displays superior scalability and prediction performance compared to state-of-the-art deep learning approaches. Additionally, we illustrate the generalization properties of Komet by showing its performance on an external dataset, and on the publicly available benchmark designed for scaffold hopping problems. Komet is available open source at https://komet.readthedocs.io and all datasets, including LCIdb, can be found at https://zenodo.org/records/10731712.
中文翻译:

大规模药物-靶标相互作用预测:使用 LCIdb 数据集的 Komet 算法
药物-靶点相互作用 (DTI) 预测算法用于药物发现过程的各个阶段。在这种情况下,诸如新治疗靶点的去孤儿化或表型筛选产生的候选药物的靶点鉴定等具体问题需要跨蛋白质和分子空间进行大规模预测。 DTI 预测在很大程度上依赖于监督学习算法,该算法使用已知的 DTI 来学习分子和蛋白质特征之间的关联,从而可以根据学习的模式预测新的相互作用。这些算法必须广泛适用才能实现可靠的预测,即使是在数据可能稀缺的蛋白质或分子空间区域。在本文中,我们解决了实现这些目标的两个关键挑战:构建大型、高质量的训练数据集和设计可扩展的预测方法,以便在如此大的数据集上进行训练。首先,我们介绍 LCIdb,这是一个精心策划的大型 DTI 数据集,广泛覆盖分子和可药物蛋白质空间。值得注意的是,LCIdb 包含的分子数量比公开的基准要多得多,扩大了分子空间的覆盖范围。其次,我们提出了 Komet(克罗内克优化方法),这是一种 DTI 预测管道,旨在实现可扩展性而不影响性能。 Komet 利用三步框架,结合了针对大型数据集定制的高效计算选择并涉及 Nyström 近似。 具体来说,Komet 采用了(分子、蛋白质)对的克罗内克相互作用模块,该模块可有效捕获 DTI 中的决定因素,其结构可降低计算复杂性和拟牛顿优化,确保模型可以处理大型训练集,而不会影响表现。我们的方法是在开源软件中实现的,利用 GPU 并行计算来提高效率。我们展示了我们的管道对各种数据集的兴趣,表明与最先进的深度学习方法相比,Komet 显示出卓越的可扩展性和预测性能。此外,我们通过展示 Komet 在外部数据集和公开数据集上的性能来说明 Komet 的泛化属性 ℒℋ
更新日期:2024-09-05
中文翻译:

大规模药物-靶标相互作用预测:使用 LCIdb 数据集的 Komet 算法
药物-靶点相互作用 (DTI) 预测算法用于药物发现过程的各个阶段。在这种情况下,诸如新治疗靶点的去孤儿化或表型筛选产生的候选药物的靶点鉴定等具体问题需要跨蛋白质和分子空间进行大规模预测。 DTI 预测在很大程度上依赖于监督学习算法,该算法使用已知的 DTI 来学习分子和蛋白质特征之间的关联,从而可以根据学习的模式预测新的相互作用。这些算法必须广泛适用才能实现可靠的预测,即使是在数据可能稀缺的蛋白质或分子空间区域。在本文中,我们解决了实现这些目标的两个关键挑战:构建大型、高质量的训练数据集和设计可扩展的预测方法,以便在如此大的数据集上进行训练。首先,我们介绍 LCIdb,这是一个精心策划的大型 DTI 数据集,广泛覆盖分子和可药物蛋白质空间。值得注意的是,LCIdb 包含的分子数量比公开的基准要多得多,扩大了分子空间的覆盖范围。其次,我们提出了 Komet(克罗内克优化方法),这是一种 DTI 预测管道,旨在实现可扩展性而不影响性能。 Komet 利用三步框架,结合了针对大型数据集定制的高效计算选择并涉及 Nyström 近似。 具体来说,Komet 采用了(分子、蛋白质)对的克罗内克相互作用模块,该模块可有效捕获 DTI 中的决定因素,其结构可降低计算复杂性和拟牛顿优化,确保模型可以处理大型训练集,而不会影响表现。我们的方法是在开源软件中实现的,利用 GPU 并行计算来提高效率。我们展示了我们的管道对各种数据集的兴趣,表明与最先进的深度学习方法相比,Komet 显示出卓越的可扩展性和预测性能。此外,我们通过展示 Komet 在外部数据集和公开数据集上的性能来说明 Komet 的泛化属性 ℒℋ































 京公网安备 11010802027423号
京公网安备 11010802027423号