当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Kinetic Diagram Analysis: A Python Library for Calculating Steady-State Observables of Biochemical Systems Analytically
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-08-19 , DOI: 10.1021/acs.jctc.4c00688 Nikolaus Carl Awtrey 1 , Oliver Beckstein 1, 2
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2024-08-19 , DOI: 10.1021/acs.jctc.4c00688 Nikolaus Carl Awtrey 1 , Oliver Beckstein 1, 2
Affiliation
|
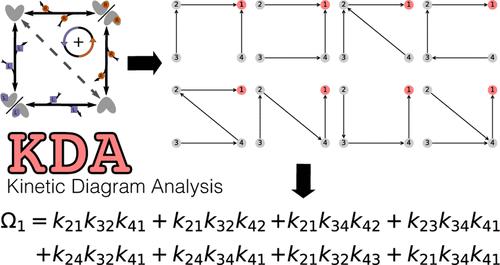
Kinetic diagrams are commonly used to represent biochemical systems in order to study phenomena such as free energy transduction and ion selectivity. While numerical methods are commonly used to analyze such kinetic networks, the diagram method by King, Altman and Hill makes it possible to construct exact algebraic expressions for steady-state observables in terms of the rate constants of the kinetic diagram. However, manually obtaining these expressions becomes infeasible for models of even modest complexity as the number of the required intermediate diagrams grows with the factorial of the number of states in the diagram. We developed Kinetic Diagram Analysis (KDA), a Python library that programmatically generates the relevant diagrams and expressions from a user-defined kinetic diagram. KDA outputs symbolic expressions for state probabilities and cycle fluxes at steady-state that can be symbolically manipulated and evaluated to quantify macroscopic system observables. We demonstrate the KDA approach for examples drawn from the biophysics of active secondary transmembrane transporters. For a generic 6-state antiporter model, we show how the introduction of a single leakage transition reduces transport efficiency by quantifying substrate turnover. We apply KDA to a real-world example, the 8-state free exchange model of the small multidrug resistance transporter EmrE of Hussey et al. (J. Gen. Physiol., 2020, 152, e201912437), where a change in transporter phenotype is achieved by biasing two different subsets of kinetic rates: alternating access and substrate unbinding rates. KDA is made available as open source software under the GNU General Public License version 3.
中文翻译:

动力学图分析:用于分析计算生化系统稳态可观察值的 Python 库
动力学图通常用于表示生化系统,以研究自由能转导和离子选择性等现象。虽然通常使用数值方法来分析此类动力学网络,但 King、Altman 和 Hill 的图方法可以根据动力学图的速率常数为稳态可观察对象构建精确的代数表达式。但是,手动获取这些表达式对于即使是中等复杂度的模型也变得不可行,因为所需的中间图的数量随着图中状态数的阶乘而增加。我们开发了动力学图分析 (KDA),这是一个 Python 库,可通过编程方式从用户定义的动力学图生成相关图表和表达式。KDA 输出稳态状态概率和周期通量的符号表达式,这些表达式可以符号操作和评估以量化宏观系统可观察对象。我们展示了 KDA 方法,例如从活性次级跨膜转运蛋白的生物物理学中得出的示例。对于通用的 6 态反向转运蛋白模型,我们展示了引入单个泄漏转变如何通过量化衬底周转来降低传输效率。我们将 KDA 应用于一个真实的例子,Hussey 等人的小型多药耐药转运蛋白 EmrE 的 8 态自由交换模型 (J. Gen. Physiol., 2020, 152, e201912437),其中转运蛋白表型的变化是通过偏置动力学速率的两个不同子集来实现的:交替访问和底物脱结合速率。KDA 在 GNU 通用公共许可证版本 3 下作为开源软件提供。
更新日期:2024-08-19
中文翻译:

动力学图分析:用于分析计算生化系统稳态可观察值的 Python 库
动力学图通常用于表示生化系统,以研究自由能转导和离子选择性等现象。虽然通常使用数值方法来分析此类动力学网络,但 King、Altman 和 Hill 的图方法可以根据动力学图的速率常数为稳态可观察对象构建精确的代数表达式。但是,手动获取这些表达式对于即使是中等复杂度的模型也变得不可行,因为所需的中间图的数量随着图中状态数的阶乘而增加。我们开发了动力学图分析 (KDA),这是一个 Python 库,可通过编程方式从用户定义的动力学图生成相关图表和表达式。KDA 输出稳态状态概率和周期通量的符号表达式,这些表达式可以符号操作和评估以量化宏观系统可观察对象。我们展示了 KDA 方法,例如从活性次级跨膜转运蛋白的生物物理学中得出的示例。对于通用的 6 态反向转运蛋白模型,我们展示了引入单个泄漏转变如何通过量化衬底周转来降低传输效率。我们将 KDA 应用于一个真实的例子,Hussey 等人的小型多药耐药转运蛋白 EmrE 的 8 态自由交换模型 (J. Gen. Physiol., 2020, 152, e201912437),其中转运蛋白表型的变化是通过偏置动力学速率的两个不同子集来实现的:交替访问和底物脱结合速率。KDA 在 GNU 通用公共许可证版本 3 下作为开源软件提供。































 京公网安备 11010802027423号
京公网安备 11010802027423号