当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
DDSBC: A Stacking Ensemble Classifier-Based Approach for Breast Cancer Drug-Pair Cell Synergy Prediction
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-08-08 , DOI: 10.1021/acs.jcim.4c01101 Aamir Mehmood 1 , Aman Chandra Kaushik 1 , Dong-Qing Wei 1
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-08-08 , DOI: 10.1021/acs.jcim.4c01101 Aamir Mehmood 1 , Aman Chandra Kaushik 1 , Dong-Qing Wei 1
Affiliation
|
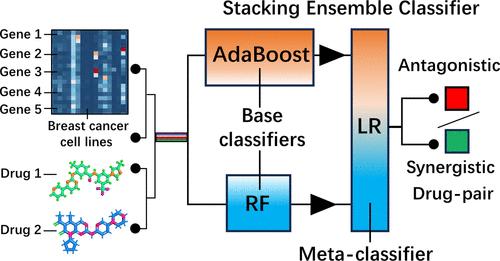
Breast cancer (BC) ranks as a leading cause of mortality among women worldwide, with incidence rates continuing to rise. The quest for effective treatments has led to the adoption of drug combination therapy, aiming to enhance drug efficacy. However, identifying synergistic drug combinations remains a daunting challenge due to the myriad of potential drug pairs. Current research leverages machine learning (ML) and deep learning (DL) models for drug-pair synergy prediction and classification. Nevertheless, these models often underperform on specific cancer types, including BC, as they are trained on data spanning various cancers without any specialization. Here, we introduce a stacking ensemble classifier, the drug–drug synergy for breast cancer (DDSBC), tailored explicitly for BC drug-pair cell synergy classification. Unlike existing models that generalize across cancer types, DDSBC is exclusively developed for BC, offering a more focused approach. Our comparative analysis against classical ML methods as well as DL models developed for drug synergy prediction highlights DDSBC’s superior performance across test and independent datasets on BC data. Despite certain metrics where other methods narrowly surpass DDSBC by 1–2%, DDSBC consistently emerges as the top-ranked model, showcasing significant differences in scoring metrics and robust performance in ablation studies. DDSBC’s performance and practicality position it as a preferred choice or an adjunctive validation tool for identifying synergistic or antagonistic drug pairs in BC, providing valuable insights for treatment strategies.
中文翻译:

DDSBC:基于堆叠集成分类器的乳腺癌药物对细胞协同预测方法
乳腺癌(BC)是全球女性死亡的主要原因,且发病率持续上升。对有效治疗的追求导致了药物联合疗法的采用,旨在提高药物疗效。然而,由于潜在的药物对无数,识别协同药物组合仍然是一项艰巨的挑战。目前的研究利用机器学习 (ML) 和深度学习 (DL) 模型进行药物对协同作用预测和分类。然而,这些模型在包括 BC 在内的特定癌症类型上通常表现不佳,因为它们是根据跨越各种癌症的数据进行训练的,而没有任何专业化。在这里,我们介绍了一种堆叠集成分类器,即乳腺癌药物协同(DDSBC),专门针对 BC 药物对细胞协同分类进行定制。与泛化癌症类型的现有模型不同,DDSBC 是专门为 BC 开发的,提供了更有针对性的方法。我们与经典 ML 方法以及为药物协同预测而开发的 DL 模型的比较分析突显了 DDSBC 在 BC 数据的测试和独立数据集上的卓越性能。尽管在某些指标上其他方法以微弱优势超过 DDSBC 1-2%,但 DDSBC 始终成为排名第一的模型,显示出评分指标的显着差异以及消融研究中的稳健表现。 DDSBC 的性能和实用性使其成为识别 BC 中协同或拮抗药物对的首选或辅助验证工具,为治疗策略提供有价值的见解。
更新日期:2024-08-08
中文翻译:

DDSBC:基于堆叠集成分类器的乳腺癌药物对细胞协同预测方法
乳腺癌(BC)是全球女性死亡的主要原因,且发病率持续上升。对有效治疗的追求导致了药物联合疗法的采用,旨在提高药物疗效。然而,由于潜在的药物对无数,识别协同药物组合仍然是一项艰巨的挑战。目前的研究利用机器学习 (ML) 和深度学习 (DL) 模型进行药物对协同作用预测和分类。然而,这些模型在包括 BC 在内的特定癌症类型上通常表现不佳,因为它们是根据跨越各种癌症的数据进行训练的,而没有任何专业化。在这里,我们介绍了一种堆叠集成分类器,即乳腺癌药物协同(DDSBC),专门针对 BC 药物对细胞协同分类进行定制。与泛化癌症类型的现有模型不同,DDSBC 是专门为 BC 开发的,提供了更有针对性的方法。我们与经典 ML 方法以及为药物协同预测而开发的 DL 模型的比较分析突显了 DDSBC 在 BC 数据的测试和独立数据集上的卓越性能。尽管在某些指标上其他方法以微弱优势超过 DDSBC 1-2%,但 DDSBC 始终成为排名第一的模型,显示出评分指标的显着差异以及消融研究中的稳健表现。 DDSBC 的性能和实用性使其成为识别 BC 中协同或拮抗药物对的首选或辅助验证工具,为治疗策略提供有价值的见解。































 京公网安备 11010802027423号
京公网安备 11010802027423号