当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Determination of Hydrogen Bonds in GROMACS: A New Implementation to Overcome Memory Limitation
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-08-09 , DOI: 10.1021/acs.jcim.3c02087 Sergey Gorelov 1, 2 , Anatoly Titov 1 , Olga Tolicheva 1 , Andrey Konevega 1, 2, 3 , Alexey Shvetsov 1, 2, 3
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-08-09 , DOI: 10.1021/acs.jcim.3c02087 Sergey Gorelov 1, 2 , Anatoly Titov 1 , Olga Tolicheva 1 , Andrey Konevega 1, 2, 3 , Alexey Shvetsov 1, 2, 3
Affiliation
|
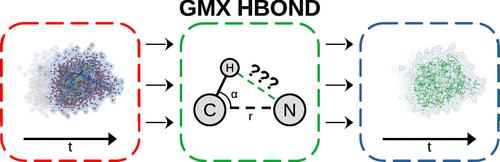
This work introduces a new faster implementation of the hydrogen bond network (complex arrangement of hydrogen bonds between or within molecules) search algorithm in biomacromolecules and their environment. Existing implementation of such an algorithm in GROMACS [Abraham et al. GROMACS 2024.2 Manual. 2024.] has limitations in the analysis of large structures and trajectories. The new implementation, in the form of a native GROMACS trajectory analysis module, allows for quick analysis of molecular dynamics trajectories without restrictions, thus overcoming the limitations of the original algorithm. The application of the developed method enabled the acquisition and analysis of hydrogen bond networks in the studied defensin-like protein Pentadiplandra brazzeana, as well as the study of hydrogen bond occupancies between the protein’s residues and water molecules. The data obtained using the new implementation coincided with the experimental data.
中文翻译:

GROMACS 中氢键的测定:克服内存限制的新实现
这项工作介绍了一种在生物大分子及其环境中更快地实现氢键网络(分子之间或分子内氢键的复杂排列)搜索算法的新方法。 GROMACS 中此类算法的现有实现 [Abraham 等人。 GROMACS 2024.2 手册。 2024 .] 在大型结构和轨迹的分析中存在局限性。新的实现以原生GROMACS轨迹分析模块的形式实现,可以不受限制地快速分析分子动力学轨迹,从而克服了原始算法的局限性。所开发的方法的应用使得能够获取和分析所研究的防御素样蛋白Pentadiplandra brazzeana中的氢键网络,以及蛋白质残基和水分子之间的氢键占据情况的研究。使用新实现获得的数据与实验数据一致。
更新日期:2024-08-09
中文翻译:

GROMACS 中氢键的测定:克服内存限制的新实现
这项工作介绍了一种在生物大分子及其环境中更快地实现氢键网络(分子之间或分子内氢键的复杂排列)搜索算法的新方法。 GROMACS 中此类算法的现有实现 [Abraham 等人。 GROMACS 2024.2 手册。 2024 .] 在大型结构和轨迹的分析中存在局限性。新的实现以原生GROMACS轨迹分析模块的形式实现,可以不受限制地快速分析分子动力学轨迹,从而克服了原始算法的局限性。所开发的方法的应用使得能够获取和分析所研究的防御素样蛋白Pentadiplandra brazzeana中的氢键网络,以及蛋白质残基和水分子之间的氢键占据情况的研究。使用新实现获得的数据与实验数据一致。

































 京公网安备 11010802027423号
京公网安备 11010802027423号