当前位置:
X-MOL 学术
›
Mol. Microbiol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Chromatin and gene regulation in archaea
Molecular Microbiology ( IF 2.6 ) Pub Date : 2024-08-03 , DOI: 10.1111/mmi.15302 Fabian Blombach 1 , Finn Werner 1
Molecular Microbiology ( IF 2.6 ) Pub Date : 2024-08-03 , DOI: 10.1111/mmi.15302 Fabian Blombach 1 , Finn Werner 1
Affiliation
|
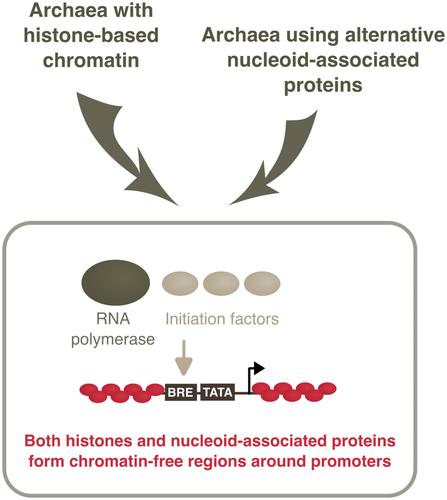
The chromatinisation of DNA by nucleoid‐associated proteins (NAPs) in archaea ‘formats’ the genome structure in profound ways, revealing both striking differences and analogies to eukaryotic chromatin. However, the extent to which archaeal NAPs actively regulate gene expression remains poorly understood. The dawn of quantitative chromatin mapping techniques and first NAP‐specific occupancy profiles in different archaea promise a more accurate view. A picture emerges where in diverse archaea with very different NAP repertoires chromatin maintains access to regulatory motifs including the gene promoter independently of transcription activity. Our re‐analysis of genome‐wide occupancy data of the crenarchaeal NAP Cren7 shows that these chromatin‐free regions are flanked by increased Cren7 binding across the transcription start site. While bacterial NAPs often form heterochromatin‐like regions across islands with xenogeneic genes that are transcriptionally silenced, there is little evidence for similar structures in archaea and data from Haloferax show that the promoters of xenogeneic genes remain accessible. Local changes in chromatinisation causing wide‐ranging effects on transcription restricted to one chromosomal interaction domain (CID) in Saccharolobus islandicus hint at a higher‐order level of organisation between chromatin and transcription. The emerging challenge is to integrate results obtained at microscale and macroscale, reconciling molecular structure and function with dynamic genome‐wide chromatin landscapes.
中文翻译:

古细菌的染色质和基因调控
古细菌中核相关蛋白 (NAP) 对 DNA 的染色质化深刻地“格式化”了基因组结构,揭示了与真核染色质的显着差异和相似之处。然而,古细菌 NAP 主动调节基因表达的程度仍然知之甚少。定量染色质作图技术的出现和不同古细菌中第一个 NAP 特异性占据图谱有望提供更准确的视图。一幅图片出现了,在具有非常不同的 NAP 库的不同古细菌中,染色质保持对包括基因启动子在内的调节基序的访问,而与转录活性无关。我们对天脑 NAP Cren7 的全基因组占用数据的重新分析表明,这些无染色质区域的两侧是跨转录起始位点的 Cren7 结合增加。虽然细菌 NAP 经常在具有转录沉默的异种基因的岛屿上形成异染色质样区域,但几乎没有证据表明古细菌中存在类似的结构,并且数据来自盐藻属表明异种基因的启动子仍然是可接近的。染色质化的局部变化对仅限于一个染色体相互作用域(CID)的转录产生广泛影响岛糖球菌暗示染色质和转录之间存在更高层次的组织。新出现的挑战是整合在微观和宏观尺度上获得的结果,使分子结构和功能与动态的全基因组染色质景观相协调。
更新日期:2024-08-03
中文翻译:

古细菌的染色质和基因调控
古细菌中核相关蛋白 (NAP) 对 DNA 的染色质化深刻地“格式化”了基因组结构,揭示了与真核染色质的显着差异和相似之处。然而,古细菌 NAP 主动调节基因表达的程度仍然知之甚少。定量染色质作图技术的出现和不同古细菌中第一个 NAP 特异性占据图谱有望提供更准确的视图。一幅图片出现了,在具有非常不同的 NAP 库的不同古细菌中,染色质保持对包括基因启动子在内的调节基序的访问,而与转录活性无关。我们对天脑 NAP Cren7 的全基因组占用数据的重新分析表明,这些无染色质区域的两侧是跨转录起始位点的 Cren7 结合增加。虽然细菌 NAP 经常在具有转录沉默的异种基因的岛屿上形成异染色质样区域,但几乎没有证据表明古细菌中存在类似的结构,并且数据来自盐藻属表明异种基因的启动子仍然是可接近的。染色质化的局部变化对仅限于一个染色体相互作用域(CID)的转录产生广泛影响岛糖球菌暗示染色质和转录之间存在更高层次的组织。新出现的挑战是整合在微观和宏观尺度上获得的结果,使分子结构和功能与动态的全基因组染色质景观相协调。






























 京公网安备 11010802027423号
京公网安备 11010802027423号