当前位置:
X-MOL 学术
›
J. Am. Chem. Soc.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Proteome Fishing for CRISPR/Cas12a-Based Orthogonal Multiplex Aptamer Sensing
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2024-07-15 , DOI: 10.1021/jacs.4c03061 Shuangqin Li 1 , Baichuan Jin 1 , Yintao Ma 1, 2 , Xu Yang 1 , Jinlong Fan 1 , Yueli Xie 1, 3 , Chenlu Xu 1, 4 , Xin Dai 1, 5 , Mengjie Wang 1, 6 , Qiqi Liu 1, 5 , Ting Fu 1, 6 , Yuan Liu 1 , Weihong Tan 1, 7
Journal of the American Chemical Society ( IF 14.4 ) Pub Date : 2024-07-15 , DOI: 10.1021/jacs.4c03061 Shuangqin Li 1 , Baichuan Jin 1 , Yintao Ma 1, 2 , Xu Yang 1 , Jinlong Fan 1 , Yueli Xie 1, 3 , Chenlu Xu 1, 4 , Xin Dai 1, 5 , Mengjie Wang 1, 6 , Qiqi Liu 1, 5 , Ting Fu 1, 6 , Yuan Liu 1 , Weihong Tan 1, 7
Affiliation
|
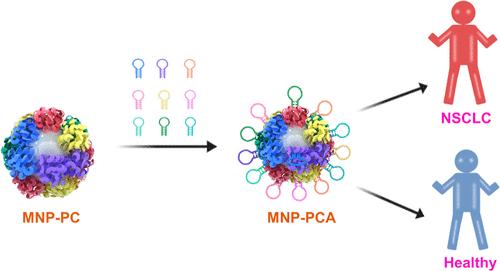
Detection of serum protein biomarkers is extremely challenging owing to the superior complexity of serum. Here, we report a method of proteome fishing from the serum. It uses a magnetic nanoparticle–protein corona and a multiplexed aptamer panel, which we incubated with the nanoparticle–protein corona for biomarker recognition. To transfer protein biomarker detection to aptamer detection, we established a CRISPR/Cas12a-based orthogonal multiplex aptamer sensing (COMPASS) platform by profiling the aptamers of protein corona with clinical nonsmall cell lung cancer (NSCLC) serum samples. Furthermore, we determined the four out of nine (FOON) panel (including HE4, NSE, AFP, and VEGF165) to be the most cost-effective and accurate panel for COMPASS in NSCLC diagnosis. The diagnostic accuracy of NSCLC by the FOON panel with internal and external cohorts was 95.56% (ROC-AUC = 99.40%) and 89.58% (ROC-AUC = 95.41%), respectively. Our developed COMPASS technology circumvents the otherwise challenging multiplexed serum protein amplification problem and avoids aptamer degradation in serum. Therefore, this novel COMPASS could lead to the development of a facile, cost-effective, intelligent, and high-throughput diagnostic platform for large-cohort cancer screening.
中文翻译:

基于 CRISPR/Cas12a 的正交多重适体传感的蛋白质组钓鱼
由于血清的高度复杂性,血清蛋白生物标志物的检测极具挑战性。在这里,我们报告了一种从血清中捕获蛋白质组的方法。它使用磁性纳米颗粒-蛋白质电晕和多重适体面板,我们将其与纳米颗粒-蛋白质电晕一起孵育以进行生物标志物识别。为了将蛋白质生物标志物检测转移到适体检测,我们通过临床非小细胞肺癌(NSCLC)血清样本对蛋白冠的适体进行分析,建立了基于CRISPR/Cas12a的正交多重适体传感(COMPASS)平台。此外,我们确定九分之四 (FOON) 组合(包括 HE4、NSE、AFP 和 VEGF165)是 COMPASS 在 NSCLC 诊断中最具成本效益和最准确的组合。 FOON 小组对内部和外部队列的 NSCLC 诊断准确率分别为 95.56% (ROC-AUC = 99.40%) 和 89.58% (ROC-AUC = 95.41%)。我们开发的 COMPASS 技术规避了原本具有挑战性的多重血清蛋白扩增问题,并避免了血清中的适体降解。因此,这种新颖的 COMPASS 可能会导致开发一种简便、经济高效、智能和高通量的诊断平台,用于大队列癌症筛查。
更新日期:2024-07-15
中文翻译:

基于 CRISPR/Cas12a 的正交多重适体传感的蛋白质组钓鱼
由于血清的高度复杂性,血清蛋白生物标志物的检测极具挑战性。在这里,我们报告了一种从血清中捕获蛋白质组的方法。它使用磁性纳米颗粒-蛋白质电晕和多重适体面板,我们将其与纳米颗粒-蛋白质电晕一起孵育以进行生物标志物识别。为了将蛋白质生物标志物检测转移到适体检测,我们通过临床非小细胞肺癌(NSCLC)血清样本对蛋白冠的适体进行分析,建立了基于CRISPR/Cas12a的正交多重适体传感(COMPASS)平台。此外,我们确定九分之四 (FOON) 组合(包括 HE4、NSE、AFP 和 VEGF165)是 COMPASS 在 NSCLC 诊断中最具成本效益和最准确的组合。 FOON 小组对内部和外部队列的 NSCLC 诊断准确率分别为 95.56% (ROC-AUC = 99.40%) 和 89.58% (ROC-AUC = 95.41%)。我们开发的 COMPASS 技术规避了原本具有挑战性的多重血清蛋白扩增问题,并避免了血清中的适体降解。因此,这种新颖的 COMPASS 可能会导致开发一种简便、经济高效、智能和高通量的诊断平台,用于大队列癌症筛查。
















































 京公网安备 11010802027423号
京公网安备 11010802027423号