当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
FCMSTrans:通过在 Transformer 框架内利用多尺度卷积和深度特征组合来准确预测与疾病相关的 nsSNP
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2024-02-13 , DOI: 10.1021/acs.jcim.3c02025
Ming Zhang 1 , Chao Gong 1 , Fang Ge 2 , Dong-Jun Yu 3
Affiliation
|
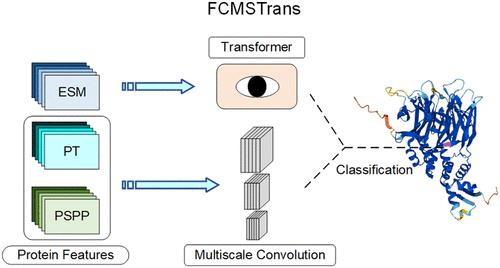
非同义单核苷酸多态性 (nsSNP) 涉及 6000 多种疾病,需要准确预测才能加快药物发现和改进疾病诊断。在这项研究中,我们提出了 FCMSTrans,一种新颖的 nsSNP 预测器,它创新性地结合了 Transformer 框架和多尺度模块来进行全面的特征提取。 FCMSTrans的独特之处在于深度特征组合策略。该策略合并了进化规模建模 (ESM) 和 ProtTrans (PT) 特征,提供了对蛋白质生化特性以及位置特异性评分矩阵、二级结构、预测相对溶剂可及性和预测无序 (PSPP) 特征的理解,这些特征是源自四种蛋白质序列和结构导向特征。该特征组合提供了涉及 nsSNP 的分子动力学的全面视图。我们的模型采用了 Transformer 跨多个层的自注意力机制,提取更高层次和抽象的表示。同时,通过多尺度卷积捕获不同级别的特征,丰富了多个梯队的特征抽象。我们与现有方法的比较分析强调了 FCMSTrans 采用的集成特征融合方法所带来的重大改进。基于不同数据集(例如 PredictSNP、MMP 和 PMD)的性能评估进一步证实了这一点,曲线下面积 (AUC) 分别为 0.869、0.819 和 0.693。此外,在第三方数据集上进行的盲测中,FCMSTrans 表现出优于当前最佳预测器 PROVEAN 的鲁棒性和优越性,取得了令人印象深刻的 AUC 分数 0.7838。 FCMSTrans 的 Python 代码可在 https://github.com/gc212/FCMSTrans 获取,供学术使用。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号