当前位置:
X-MOL 学术
›
Comput. Struct. Biotechnol. J.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
AcrIIA4 和 AcrIIA2 诱导的 SpyCas9 构象可塑性:来自分子动力学模拟的见解
Computational and Structural Biotechnology Journal ( IF 4.4 ) Pub Date : 2023-12-27 , DOI: 10.1016/j.csbj.2023.12.030 Shuixiu Wen 1 , Yuxin Zhao 1 , Xinyu Qi 1 , Mingzhu Cai 1 , Kaisheng Huang 1 , Hui Liu 1 , De-Xin Kong 1
Affiliation
|
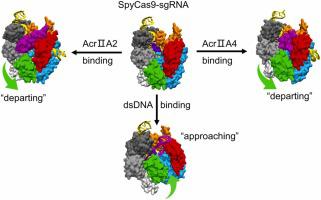
CRISPR-Cas9 系统构成细菌适应性免疫系统,可防止噬菌体感染。噬菌体编码抗 CRISPR 蛋白 (Acrs),可减轻细菌免疫反应。然而,从分子角度来看,它们的抑制作用的结构基础仍然难以捉摸。在本研究中,通过微秒原子分子动力学模拟,我们展示了化脓性链球菌Cas9(SpyCas9)在与AcrIIA4和AcrIIA2相互作用过程中的显着灵活性及其构象适应性。具体来说,我们证明 AcrIIA4 和 AcrIIA2 与 SpyCas9 的结合会诱导构象重排,导致核酸酶和切割位点之间的空间分离,从而使核酸内切酶失活。这种分离破坏了原型间隔区相邻基序识别和核酸酶结构域之间的信号传输,从而阻碍了双链 DNA 的有效处理。模拟还揭示了 AcrIIA4 和 AcrIIA2 导致 SpyCas9 的不同结构变化。我们的研究阐明了 AcrIIA4 和 AcrIIA2 抑制 SpyCas9 的精确机制,从而为更准确地控制基因组编辑提供了新的可能性。

"点击查看英文标题和摘要"





















































 京公网安备 11010802027423号
京公网安备 11010802027423号