Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Kinetic FRET Assay to Measure Binding-Induced Conformational Changes of Nucleic Acids
ACS Sensors ( IF 8.2 ) Pub Date : 2023-12-07 , DOI: 10.1021/acssensors.3c01527 R Anahi Higuera-Rodriguez 1, 2 , Mareike C De Pascali 1, 2 , Masood Aziz 1, 3 , Michael Sattler 1, 3 , Ulrich Rant 2 , Wolfgang Kaiser 2
ACS Sensors ( IF 8.2 ) Pub Date : 2023-12-07 , DOI: 10.1021/acssensors.3c01527 R Anahi Higuera-Rodriguez 1, 2 , Mareike C De Pascali 1, 2 , Masood Aziz 1, 3 , Michael Sattler 1, 3 , Ulrich Rant 2 , Wolfgang Kaiser 2
Affiliation
|
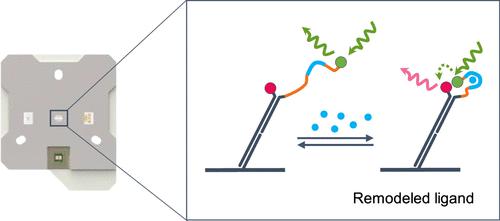
The interaction of small molecules or proteins with RNA or DNA often involves changes in the nucleic acid (NA) folding and structure. A biophysical characterization of these processes helps us to understand the underlying molecular mechanisms. Here, we propose kinFRET (kinetics Förster resonance energy transfer), a real-time ensemble FRET methodology to measure binding and folding kinetics. With kinFRET, the kinetics of conformational changes of NAs (DNA or RNA) upon analyte binding can be directly followed via a FRET signal using a chip-based biosensor. We demonstrate the utility of this approach with two representative examples. First, we monitored the conformational changes of different formats of an aptamer (MN19) upon interaction with small-molecule analytes. Second, we characterized the binding kinetics of RNA recognition by tandem K homology (KH) domains of the human insulin-like growth factor II mRNA-binding protein 3 (IMP3), which reveals distinct kinetic contributions of the two KH domains. Our data demonstrate that kinFRET is well suited to study the kinetics and conformational changes of NA–analyte interactions.
中文翻译:

动态 FRET 测定法测量结合诱导的核酸构象变化
小分子或蛋白质与 RNA 或 DNA 的相互作用通常涉及核酸 (NA) 折叠和结构的变化。这些过程的生物物理表征有助于我们了解潜在的分子机制。在这里,我们提出了 kinFRET(动力学福斯特共振能量转移),这是一种实时集成 FRET 方法,用于测量结合和折叠动力学。借助 kinFRET,可以使用基于芯片的生物传感器通过 FRET 信号直接跟踪分析物结合时 NA(DNA 或 RNA)构象变化的动力学。我们通过两个代表性示例展示了这种方法的实用性。首先,我们监测了不同形式的适体 (MN19) 在与小分子分析物相互作用时的构象变化。其次,我们通过人胰岛素样生长因子 II mRNA 结合蛋白 3 (IMP3) 的串联 K 同源 (KH) 结构域表征了 RNA 识别的结合动力学,这揭示了两个 KH 结构域的独特动力学贡献。我们的数据表明 kinFRET 非常适合研究 NA-分析物相互作用的动力学和构象变化。
更新日期:2023-12-07
中文翻译:

动态 FRET 测定法测量结合诱导的核酸构象变化
小分子或蛋白质与 RNA 或 DNA 的相互作用通常涉及核酸 (NA) 折叠和结构的变化。这些过程的生物物理表征有助于我们了解潜在的分子机制。在这里,我们提出了 kinFRET(动力学福斯特共振能量转移),这是一种实时集成 FRET 方法,用于测量结合和折叠动力学。借助 kinFRET,可以使用基于芯片的生物传感器通过 FRET 信号直接跟踪分析物结合时 NA(DNA 或 RNA)构象变化的动力学。我们通过两个代表性示例展示了这种方法的实用性。首先,我们监测了不同形式的适体 (MN19) 在与小分子分析物相互作用时的构象变化。其次,我们通过人胰岛素样生长因子 II mRNA 结合蛋白 3 (IMP3) 的串联 K 同源 (KH) 结构域表征了 RNA 识别的结合动力学,这揭示了两个 KH 结构域的独特动力学贡献。我们的数据表明 kinFRET 非常适合研究 NA-分析物相互作用的动力学和构象变化。































 京公网安备 11010802027423号
京公网安备 11010802027423号