当前位置:
X-MOL 学术
›
Fish Fish.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Trees for fishes: The neglected role for phylogenetic comparative methods in fisheries science
Fish and Fisheries ( IF 5.6 ) Pub Date : 2023-11-16 , DOI: 10.1111/faf.12800 James T. Thorson 1
Fish and Fisheries ( IF 5.6 ) Pub Date : 2023-11-16 , DOI: 10.1111/faf.12800 James T. Thorson 1
Affiliation
|
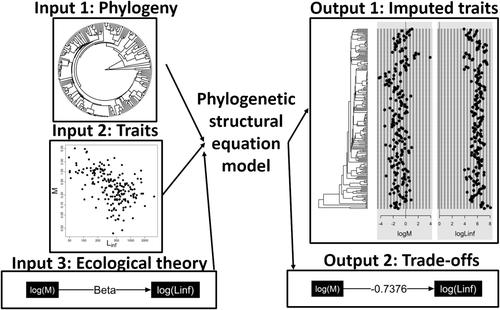
Fisheries scientists compare processes among species to estimate species productivity, management reference points, and climate sensitivities. Ecologists have developed “phylogenetic comparative methods” (PCMs) to address these questions, but there is surprisingly little application of PCM within fisheries science. Here, I bridge this gap by introducing PCM (including Brownian motion, Ornstein–Uhlenbeck, and Pagel's kappa and lambda models for species covariance), thereby showing that PCM generalizes the nested taxonomic random effects that are commonly used in fisheries science. I next summarize phylogenetic structural equation models (PSEMs), which extend the linear models that are commonly used in fisheries. Finally, I re-analyse a high-quality database used to predict mortality rates from longevity and/or growth parameters. I specifically propose a PSEM that reverts to a longevity-based prediction when longevity information is available but uses phylogenetic corrected growth parameters otherwise. Using this single PSEM replaces the common practice of fitting and predicting using separate linear models depending upon what data are available for a given species. Cross-validation suggests that the relationship between log-mortality rate and longevity does not vary based on phylogeny, and therefore, linear models and PSEM both explain 82% of variance when longevity is available. When longevity is unavailable, by contrast, the linear model explains only 37% of variance while the PSEM explains 52% of variance, where this gain occurs from conditioning predictions on phylogenetic similarities. I therefore conclude that PCM and PSEM provide a general and user-friendly replacement for linear models and can improve performance for fisheries meta-analyses that are used for fisheries management applications.
中文翻译:

鱼类之树:系统发育比较方法在渔业科学中被忽视的作用
渔业科学家比较物种之间的过程,以估计物种生产力、管理参考点和气候敏感性。生态学家开发了“系统发育比较方法”(PCM)来解决这些问题,但令人惊讶的是 PCM 在渔业科学中的应用很少。在这里,我通过引入 PCM(包括布朗运动、Ornstein-Uhlenbeck 以及用于物种协方差的 Pagel 的 kappa 和 lambda 模型)来弥补这一差距,从而表明 PCM 概括了渔业科学中常用的嵌套分类随机效应。接下来我总结了系统发育结构方程模型(PSEM),它扩展了渔业中常用的线性模型。最后,我重新分析了一个用于根据寿命和/或生长参数预测死亡率的高质量数据库。我特别提出了一个 PSEM,当寿命信息可用时,它会恢复到基于寿命的预测,但否则使用系统发育校正的生长参数。使用这个单一的 PSEM 取代了根据给定物种可用的数据使用单独的线性模型进行拟合和预测的常见做法。交叉验证表明,对数死亡率和寿命之间的关系不会因系统发育而变化,因此,当寿命可用时,线性模型和 PSEM 都可以解释 82% 的方差。相比之下,当寿命不可用时,线性模型仅解释 37% 的方差,而 PSEM 解释 52% 的方差,这种增益来自对系统发育相似性的条件预测。因此,我得出的结论是,PCM 和 PSEM 为线性模型提供了通用且用户友好的替代方案,并且可以提高用于渔业管理应用的渔业荟萃分析的性能。
更新日期:2023-11-16
中文翻译:

鱼类之树:系统发育比较方法在渔业科学中被忽视的作用
渔业科学家比较物种之间的过程,以估计物种生产力、管理参考点和气候敏感性。生态学家开发了“系统发育比较方法”(PCM)来解决这些问题,但令人惊讶的是 PCM 在渔业科学中的应用很少。在这里,我通过引入 PCM(包括布朗运动、Ornstein-Uhlenbeck 以及用于物种协方差的 Pagel 的 kappa 和 lambda 模型)来弥补这一差距,从而表明 PCM 概括了渔业科学中常用的嵌套分类随机效应。接下来我总结了系统发育结构方程模型(PSEM),它扩展了渔业中常用的线性模型。最后,我重新分析了一个用于根据寿命和/或生长参数预测死亡率的高质量数据库。我特别提出了一个 PSEM,当寿命信息可用时,它会恢复到基于寿命的预测,但否则使用系统发育校正的生长参数。使用这个单一的 PSEM 取代了根据给定物种可用的数据使用单独的线性模型进行拟合和预测的常见做法。交叉验证表明,对数死亡率和寿命之间的关系不会因系统发育而变化,因此,当寿命可用时,线性模型和 PSEM 都可以解释 82% 的方差。相比之下,当寿命不可用时,线性模型仅解释 37% 的方差,而 PSEM 解释 52% 的方差,这种增益来自对系统发育相似性的条件预测。因此,我得出的结论是,PCM 和 PSEM 为线性模型提供了通用且用户友好的替代方案,并且可以提高用于渔业管理应用的渔业荟萃分析的性能。
















































 京公网安备 11010802027423号
京公网安备 11010802027423号