当前位置:
X-MOL 学术
›
ACS Synth. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Highly Accurate Sequence- and Position-Independent Error Profiling of DNA Synthesis and Sequencing
ACS Synthetic Biology ( IF 3.7 ) Pub Date : 2023-11-14 , DOI: 10.1021/acssynbio.3c00308 Huiran Yeom 1 , Namphil Kim 2 , Amos Chungwon Lee 3 , Jinhyun Kim 2 , Hamin Kim 4 , Hansol Choi 5 , Seo Woo Song 6 , Sunghoon Kwon 2, 4, 5 , Yeongjae Choi 7
ACS Synthetic Biology ( IF 3.7 ) Pub Date : 2023-11-14 , DOI: 10.1021/acssynbio.3c00308 Huiran Yeom 1 , Namphil Kim 2 , Amos Chungwon Lee 3 , Jinhyun Kim 2 , Hamin Kim 4 , Hansol Choi 5 , Seo Woo Song 6 , Sunghoon Kwon 2, 4, 5 , Yeongjae Choi 7
Affiliation
|
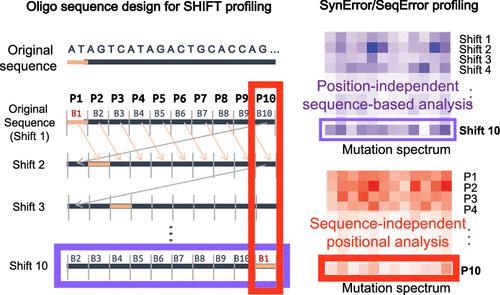
A comprehensive error analysis of DNA-stored data during processing, such as DNA synthesis and sequencing, is crucial for reliable DNA data storage. Both synthesis and sequencing errors depend on the sequence and the transition of bases of nucleotides; ignoring either one of the error sources leads to technical challenges in minimizing the error rate. Here, we present a methodology and toolkit that utilizes an oligonucleotide library generated from a 10-base-shifted sequence array, which is individually labeled with unique molecular identifiers, to delineate and profile DNA synthesis and sequencing errors simultaneously. This methodology enables position- and sequence-independent error profiling of both DNA synthesis and sequencing. Using this toolkit, we report base transitional errors in both synthesis and sequencing in general DNA data storage as well as degenerate-base-augmented DNA data storage. The methodology and data presented will contribute to the development of DNA sequence designs with minimal error.
中文翻译:

DNA 合成和测序的高精度序列和位置无关错误分析
在处理过程中对 DNA 存储的数据进行全面的错误分析,例如 DNA 合成和测序,对于可靠的 DNA 数据存储至关重要。合成和测序错误都取决于核苷酸碱基的序列和转变;忽略任何一个错误源都会导致在最小化错误率方面面临技术挑战。在这里,我们提出了一种方法和工具包,该方法和工具包利用从 10 个碱基移位序列阵列生成的寡核苷酸文库,该序列阵列用独特的分子标识符单独标记,以同时描述和分析 DNA 合成和测序错误。该方法能够对 DNA 合成和测序进行不依赖于位置和序列的错误分析。使用该工具包,我们报告了一般 DNA 数据存储以及简并碱基增强 DNA 数据存储中合成和测序中的碱基过渡错误。所提供的方法和数据将有助于开发误差最小的 DNA 序列设计。
更新日期:2023-11-14
中文翻译:

DNA 合成和测序的高精度序列和位置无关错误分析
在处理过程中对 DNA 存储的数据进行全面的错误分析,例如 DNA 合成和测序,对于可靠的 DNA 数据存储至关重要。合成和测序错误都取决于核苷酸碱基的序列和转变;忽略任何一个错误源都会导致在最小化错误率方面面临技术挑战。在这里,我们提出了一种方法和工具包,该方法和工具包利用从 10 个碱基移位序列阵列生成的寡核苷酸文库,该序列阵列用独特的分子标识符单独标记,以同时描述和分析 DNA 合成和测序错误。该方法能够对 DNA 合成和测序进行不依赖于位置和序列的错误分析。使用该工具包,我们报告了一般 DNA 数据存储以及简并碱基增强 DNA 数据存储中合成和测序中的碱基过渡错误。所提供的方法和数据将有助于开发误差最小的 DNA 序列设计。































 京公网安备 11010802027423号
京公网安备 11010802027423号