当前位置:
X-MOL 学术
›
J. Chem. Inf. Model.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
使用分子动力学模拟破译 SIRT6 的变构激活机制
Journal of Chemical Information and Modeling ( IF 5.6 ) Pub Date : 2023-08-31 , DOI: 10.1021/acs.jcim.3c00227
Zhiyuan Zhao 1 , Jintong Du 1, 2 , Yu Du 1 , Yuan Gao 1 , Mingxuan Yu 1 , Yingkai Zhang 3, 4 , Hao Fang 1 , Xuben Hou 1
Affiliation
|
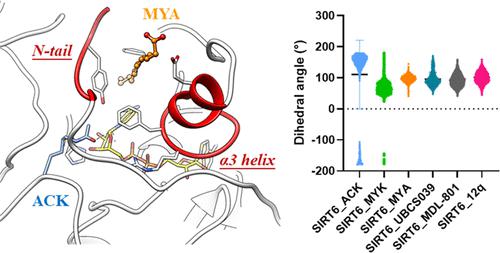
作为组蛋白脱乙酰酶蛋白家族的一员,NAD +依赖的SIRT6在维持基因组稳定性和调节细胞代谢方面发挥着重要作用。有趣的是,SIRT6被发现相对于脱乙酰化更倾向于水解长链脂肪酰基,并且它可以被脂肪酸激活。然而,SIRT6 识别不同底物并被小分子激活剂激活的机制仍不清楚。在这项研究中,我们进行了广泛的分子动力学模拟,以阐明这些机制。我们的结果表明,肉豆蔻酰化底物的结合稳定了 NAD +的催化有利构象,而乙酰基赖氨酸底物的结合导致 SIRT6 中 NAD +的松散结合。基于这些观察,我们提出了肉豆蔻酸合理的变构结合模式,该模式可以通过稳定NAD +与His131以及乙酰化底物的结合来增强SIRT6的催化活性。此外,我们的分子动力学模拟表明,合成的 SIRT6 激活剂(例如 UBCS039、MDL-801 和 12q)可阻止 NAD +中核糖的翻转,因此可以以类似于脂肪酸的方式稳定底物 -NAD + -His131 相互作用。总之,我们新提出的 SIRT6 激活机制强调了蛋白质-底物相互作用的重要性,这将有助于新 SIRT6 激活剂的合理设计。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号