当前位置:
X-MOL 学术
›
Neuroinformatics
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
3D Atlas 空间中 2D 组织学小鼠大脑图像的定位和配准
Neuroinformatics ( IF 2.7 ) Pub Date : 2023-06-26 , DOI: 10.1007/s12021-023-09632-8 Maryam Sadeghi 1 , Arnau Ramos-Prats 2 , Pedro Neto 3 , Federico Castaldi 2 , Devin Crowley 4 , Pawel Matulewicz 2 , Enrica Paradiso 5 , Wolfgang Freysinger 6 , Francesco Ferraguti 2 , Georg Goebel 1
Affiliation
|
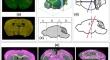
为了准确探索大脑神经回路的解剖组织,将实验大脑数据映射到标准化坐标系统至关重要。研究二维组织学小鼠脑切片仍然是许多实验室的标准程序。绘制这些二维大脑切片的地图具有挑战性;由于在标准制备和切片过程中引入的变形、伪影和倾斜角度。此外,实验小鼠脑切片的分析可能高度依赖于操作人员的专业水平。在这里,我们提出了一种用于精确小鼠大脑图像分析 (AMBIA) 的计算工具,可以在最少的人为干预的情况下将 2D 小鼠大脑切片映射到 3D 大脑模型上。 AMBIA采用模块化设计,包括本地化模块和注册模块。定位模块是一个基于深度学习的管道,可定位 3D Allen Brain Atlas 中的单个 2D 切片并生成相应的图集平面。配准模块基于 Ardent python 包构建,该包在大脑切片与其相应的图集之间执行可变形的 2D 配准。通过将 AMBIA 在本地化和注册方面的表现与人类评分进行比较,我们证明它的表现达到了人类专家的水平。 AMBIA 提供了一种直观且高效的方法,可将实验性 2D 小鼠大脑图像准确配准到 3D 数字小鼠大脑图谱。我们的工具提供图形用户界面,旨在供具有最少编程知识的研究人员使用。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号