当前位置:
X-MOL 学术
›
J. Biophotonics
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
拉曼光谱结合变分自动编码器和深度学习对病原菌进行分类
Journal of Biophotonics ( IF 2.0 ) Pub Date : 2022-12-15 , DOI: 10.1002/jbio.202200270 Bo Liu 1, 2 , Kunxiang Liu 1, 2 , Jide Sun 3 , Lindong Shang 1, 2 , Qingxiang Yang 4 , Xueping Chen 5 , Bei Li 1, 2
Journal of Biophotonics ( IF 2.0 ) Pub Date : 2022-12-15 , DOI: 10.1002/jbio.202200270 Bo Liu 1, 2 , Kunxiang Liu 1, 2 , Jide Sun 3 , Lindong Shang 1, 2 , Qingxiang Yang 4 , Xueping Chen 5 , Bei Li 1, 2
Affiliation
|
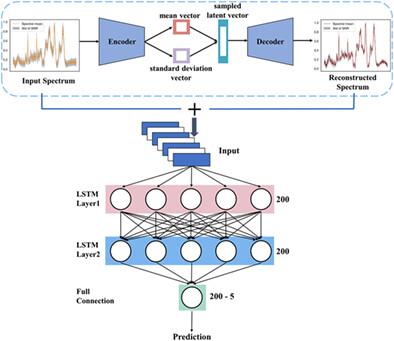
病原体的快速和早期鉴定对于指导抗生素治疗至关重要。拉曼光谱作为一种无创诊断技术,可以快速准确地检测病原体。单细胞的拉曼光谱作为细胞的“指纹”,揭示其代谢特征。通过结合拉曼光谱和深度学习,可以实现病原体的快速识别。传统的分类技术通常需要大量数据进行训练,这会花费大量时间来收集拉曼光谱。对于难以培养的微量样品和菌株,很难提供准确的分类模型。为了减少采集的样本数量并提高分类模型的准确性,一种新的结合拉曼光谱的病原体检测方法,本文提出了变分自动编码器 (VAE) 和长短期记忆网络 (LSTM)。我们收集病原体的拉曼信号并将其输入到 VAE 进行训练。VAE会产生大量与真实光谱无法区分的拉曼光谱数据,信噪比高于真实光谱。这些光谱与真实光谱一起输入LSTM进行训练,得到良好的分类模型。实验结果表明,该方法不仅将病原体分类的平均准确率提高到96.9%,而且将采集的拉曼光谱数量从1000个减少到200个。采用该技术,采集的拉曼光谱数量可以大大减少,以便快速鉴定难以培养或追踪的菌株。本文提出了长短期记忆网络(LSTM)。我们收集病原体的拉曼信号并将其输入到 VAE 进行训练。VAE会产生大量与真实光谱无法区分的拉曼光谱数据,信噪比高于真实光谱。这些光谱与真实光谱一起输入LSTM进行训练,得到良好的分类模型。实验结果表明,该方法不仅将病原体分类的平均准确率提高到96.9%,而且将采集的拉曼光谱数量从1000个减少到200个。采用该技术,采集的拉曼光谱数量可以大大减少,以便快速鉴定难以培养或追踪的菌株。本文提出了长短期记忆网络(LSTM)。我们收集病原体的拉曼信号并将其输入到 VAE 进行训练。VAE会产生大量与真实光谱无法区分的拉曼光谱数据,信噪比高于真实光谱。这些光谱与真实光谱一起输入LSTM进行训练,得到良好的分类模型。实验结果表明,该方法不仅将病原体分类的平均准确率提高到96.9%,而且将采集的拉曼光谱数量从1000个减少到200个。采用该技术,采集的拉曼光谱数量可以大大减少,以便快速鉴定难以培养或追踪的菌株。

"点击查看英文标题和摘要"
更新日期:2022-12-15


"点击查看英文标题和摘要"















































 京公网安备 11010802027423号
京公网安备 11010802027423号