当前位置:
X-MOL 学术
›
Nat. Commun.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
用于基准祖先序列重建的实验系统发育。
Nature Communications ( IF 14.7 ) Pub Date : 2016-09-15 , DOI: 10.1038/ncomms12847
Ryan N Randall 1 , Caelan E Radford 1 , Kelsey A Roof 1 , Divya K Natarajan 1 , Eric A Gaucher 1, 2
Affiliation
|
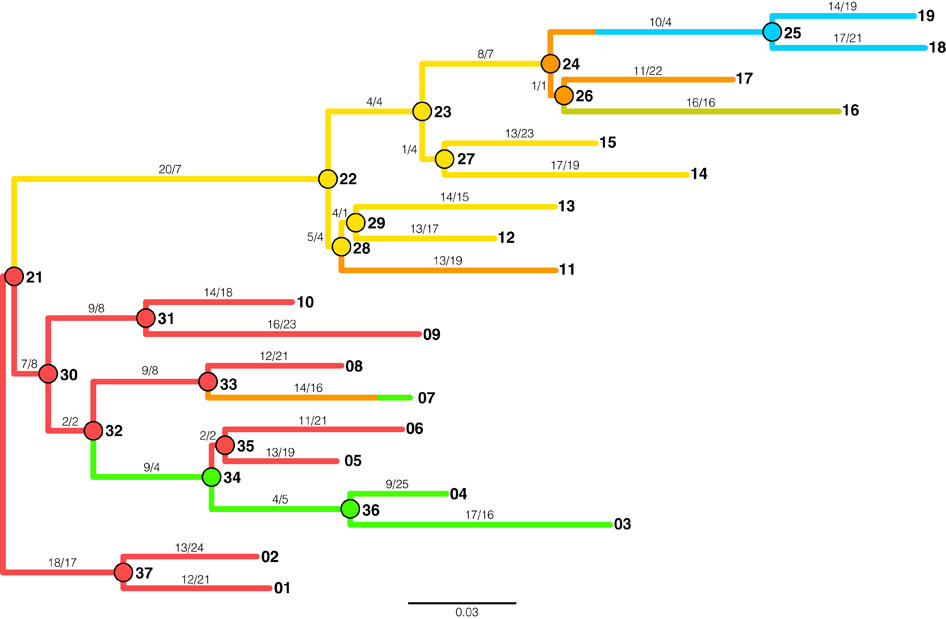
祖先序列重建(ASR)是一种仍在新兴的方法,它揭示了分子进化的许多关键机制。该方法的一个批评是无法在生物环境(而不是计算机模拟)中验证其算法。在这里,我们使用单个红色荧光蛋白的基因构建了一个实验系统发育学来解决这一批评。进化的系统发育由 19 个操作分类单元(叶)和 17 个祖先分叉(节点)组成,显示出多种荧光表型。然后,这 19 个叶子作为“现代”序列,我们使用各种算法对其进行 ASR 分析,并以已知的祖先基因型和祖先表型为基准。我们确认计算机模拟显示所有算法都能高精度地推断出古代序列,但我们也揭示了由错误推断的序列编码的表型的巨大差异。具体来说,结合速率变化的贝叶斯方法在表型准确性方面显着优于最大简约标准。现有序列的二次采样对祖先序列的推断影响较小。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号