当前位置:
X-MOL 学术
›
Ecol. Evol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
DNA 提取过程中的重复子样本揭示 Malaise trap DNA 元条形码的多样性估计增加
Ecology and Evolution ( IF 2.3 ) Pub Date : 2022-11-27 , DOI: 10.1002/ece3.9502
Vera M A Zizka 1 , Matthias F Geiger 1 , Thomas Hörren 2 , Ameli Kirse 1 , Niklas W Noll 1 , Livia Schäffler 1 , Alice M Scherges 1 , Martin Sorg 2
Affiliation
|
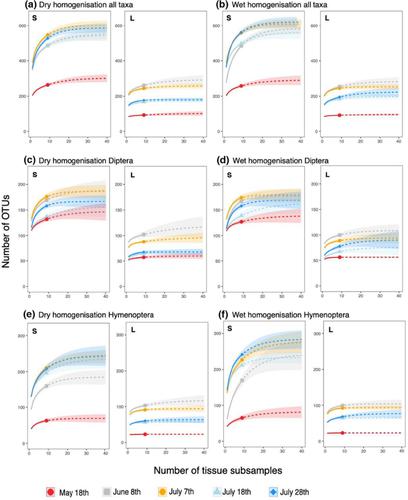
随着DNA元条形码在生物多样性评估中的应用不断增加,各种实验室方案已得到优化,其进一步评估是当前研究的主题。大量样品的均质化以及随后从受损组织的子样品中提取 DNA 是元条形码过程的常见第一阶段。这可以使用浸泡在储存固定剂(例如乙醇)中的样品材料(此处称为“湿”处理)或来自干燥的个体(“干”)来进行。然而,仍然不确定在均质化过程中是否能确保试管内 DNA 的完美混合和均匀分布,以及如果在下游元条形码工作流程中仅处理一小部分被破坏的样品,则不完全混合和由此产生的组织成分变化会在多大程度上影响多样性评估。在这里,我们研究了潮湿和干燥条件下的均质化效率,并测试了破坏组织成分的变化如何影响复杂节肢动物样本的多样性评估。我们考虑了 Malaise trap 批量样品的五个时间间隔,并在两种处理中处理了九个不同的均质组织子样品(每个 20 毫克)。结果表明,干燥材料的多样性评估更加一致,但代价是处理时间更长。两种方法都检测到了相当的 OTU 多样性,并在单次组织提取中揭示了相似的分类群组成。随着 DNA 提取过程中组织子样本数量的增加,两种方法的 OTU 多样性都增加,特别是对于夏季获得的高度多样化的样本。在这里,特别是小型和低生物量类群的检测有所增加。 因此,元条形码协议中的多个子样本的处理可以成为增强多样性估计和抵消生物多样性评估中的分类偏差的有用程序。然而,该过程会导致更高的成本和时间投入,并且需要根据具体项目的前景考虑在大规模生物多样性评估中的应用,例如在监测计划中的应用。

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号