当前位置:
X-MOL 学术
›
ACS Chem. Biol.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
Structures of Methanomethylophilus alvus Pyrrolysine tRNA-Synthetases Support the Need for De Novo Selections When Altering the Substrate Specificity
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2022-11-17 , DOI: 10.1021/acschembio.2c00640 Ilana Gottfried-Lee 1 , John J Perona 2 , P Andrew Karplus 1 , Ryan A Mehl 1 , Richard B Cooley 1
ACS Chemical Biology ( IF 3.5 ) Pub Date : 2022-11-17 , DOI: 10.1021/acschembio.2c00640 Ilana Gottfried-Lee 1 , John J Perona 2 , P Andrew Karplus 1 , Ryan A Mehl 1 , Richard B Cooley 1
Affiliation
|
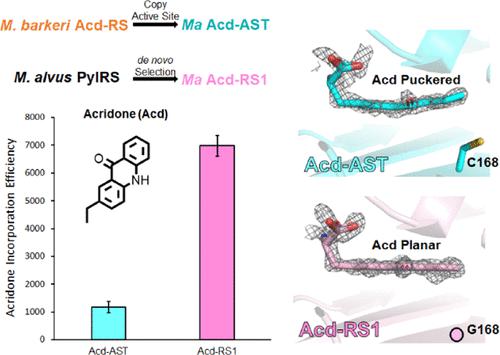
A recently developed genetic code expansion (GCE) platform based on the pyrrolysine amino-acyl tRNA synthetase (PylRS)/tRNAPyl pair from Methanomethylophilus alvus(Ma) has improved solubility and lower susceptibility to proteolysis compared with the homologous and commonly used Methanosarcina barkeri(Mb) and M. mazei(Mm) PylRS GCE platforms. We recently created two new Ma PylRS variants for the incorporation of the fluorescent amino acid, acridonyl-alanine (Acd), into proteins at amber codons: one based on “transplanting” active site mutations from an established high-efficiency Mb PylRS and one that was de novo selected from a library of mutants. Here, we present the crystal structures of these two Ma PylRS variants with Acd/ATP bound to understand why the “active site transplant” variant (Acd-AST) displayed 6-fold worse Acd incorporation efficiency than the de novo selected PylRS (called Acd-RS1). The structures reveal that the Acd-AST binding pocket is too small and binds the three-ring aromatic Acd in a distorted conformation, whereas the more spacious Acd-RS1 active site binds Acd in a relaxed, planar conformation stabilized by a network of solvent-mediated hydrogen bonds. The poor performance of the AST enzyme is ascribed to a shift in the Ma PylRS β-sheet framework relative to that of the Mb enzyme. This illustrates a general reason why “active site transplantation” may not succeed in creating efficient Ma PylRSs for other noncanonical amino acids. This work also provides structural details that will help guide the development of future Ma PylRS/tRNAPyl GCE systems via de novo selection or directed evolution methods.
中文翻译:

嗜甲烷菌吡咯赖氨酸 tRNA 合成酶的结构支持改变底物特异性时需要从头选择
最近开发的基于来自Methanomethylophilus alvus (Ma)的吡咯赖氨酸氨基酰基 tRNA 合成酶 (PylRS)/tRNA Pyl对的遗传密码扩展 (GCE) 平台与同源且常用的Methanosarcina barkeri相比,具有更高的溶解度和更低的蛋白水解敏感性。 Mb)和M. mazei (Mm) PylRS GCE 平台。我们最近创建了两种新的Ma PylRS 变体,用于将荧光氨基酸吖啶酮丙氨酸 (Acd) 掺入琥珀密码子的蛋白质中:一种基于从已建立的高效Mb PylRS 中“移植”活性位点突变,另一种基于从已建立的高效 Mb PylRS 中“移植”活性位点突变是从突变体库中从头选择的。在这里,我们展示了这两种Ma PylRS 变体与 Acd/ATP 结合的晶体结构,以了解为什么“活性位点移植”变体 (Acd-AST) 的 Acd 掺入效率比从头选择的 PylRS(称为 Acd)差 6 倍。 -RS1)。结构表明,Acd-AST 结合口袋太小,以扭曲的构象结合三环芳香 Acd,而更宽敞的 Acd-RS1 活性位点以松弛的平面构象结合,由溶剂网络稳定。介导的氢键。 AST 酶的较差性能归因于Ma PylRS β-折叠框架相对于Mb酶的移动。这说明了“活性位点移植”可能无法成功为其他非规范氨基酸创建有效的Ma PylRS 的一般原因。 这项工作还提供了结构细节,有助于通过从头选择或定向进化方法指导未来Ma PylRS/tRNA Pyl GCE 系统的开发。
更新日期:2022-11-17
中文翻译:

嗜甲烷菌吡咯赖氨酸 tRNA 合成酶的结构支持改变底物特异性时需要从头选择
最近开发的基于来自Methanomethylophilus alvus (Ma)的吡咯赖氨酸氨基酰基 tRNA 合成酶 (PylRS)/tRNA Pyl对的遗传密码扩展 (GCE) 平台与同源且常用的Methanosarcina barkeri相比,具有更高的溶解度和更低的蛋白水解敏感性。 Mb)和M. mazei (Mm) PylRS GCE 平台。我们最近创建了两种新的Ma PylRS 变体,用于将荧光氨基酸吖啶酮丙氨酸 (Acd) 掺入琥珀密码子的蛋白质中:一种基于从已建立的高效Mb PylRS 中“移植”活性位点突变,另一种基于从已建立的高效 Mb PylRS 中“移植”活性位点突变是从突变体库中从头选择的。在这里,我们展示了这两种Ma PylRS 变体与 Acd/ATP 结合的晶体结构,以了解为什么“活性位点移植”变体 (Acd-AST) 的 Acd 掺入效率比从头选择的 PylRS(称为 Acd)差 6 倍。 -RS1)。结构表明,Acd-AST 结合口袋太小,以扭曲的构象结合三环芳香 Acd,而更宽敞的 Acd-RS1 活性位点以松弛的平面构象结合,由溶剂网络稳定。介导的氢键。 AST 酶的较差性能归因于Ma PylRS β-折叠框架相对于Mb酶的移动。这说明了“活性位点移植”可能无法成功为其他非规范氨基酸创建有效的Ma PylRS 的一般原因。 这项工作还提供了结构细节,有助于通过从头选择或定向进化方法指导未来Ma PylRS/tRNA Pyl GCE 系统的开发。

































 京公网安备 11010802027423号
京公网安备 11010802027423号