当前位置:
X-MOL 学术
›
J. Chem. Theory Comput.
›
论文详情
Our official English website, www.x-mol.net, welcomes your
feedback! (Note: you will need to create a separate account there.)
双链 DNA 构象和弹性特性的精确序列依赖粗粒度模型
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2022-04-08 , DOI: 10.1021/acs.jctc.2c00138 Salvatore Assenza 1 , Rubén Pérez 1
Journal of Chemical Theory and Computation ( IF 5.7 ) Pub Date : 2022-04-08 , DOI: 10.1021/acs.jctc.2c00138 Salvatore Assenza 1 , Rubén Pérez 1
Affiliation
|
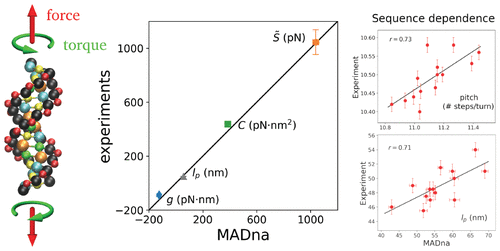
我们介绍了 MADna,这是一种依赖于序列的双链 DNA (dsDNA) 粗粒度模型,其中每个核苷酸由三个分别位于糖、碱基和磷酸基团的珠子描述。通过考虑键合相互作用的阶跃参数化来包括序列依赖性,对其进行调整以重现从文献中详尽的原子模拟获得的关键可观察量的值。该模型的预测以一组独立的全原子模拟为基准,表明它以高保真度捕获了构象和弹性特征的序列依赖性,超出了其公式中考虑的单个步骤。对于序列平均和序列相关的构象和弹性特征,包括拉伸和扭转模量、扭曲-拉伸和扭曲-弯曲耦合、持久长度和螺旋间距,发现与实验非常吻合。总体而言,对于检查的数量,该模型具有与原子模拟相当的精度,因此为单分子实验的合理化和涉及 dsDNA 的细胞过程的研究提供了可靠的粗粒度描述。由于其公式的简单性,MADna 可以直接包含在常见的仿真引擎中。特别是,LAMMPS 模型的实现可在在线存储库中获得,以简化其在 DNA 研究社区中的使用。包括拉伸和扭转模量、扭转-拉伸和扭转-弯曲耦合、持续长度和螺距。总体而言,对于检查的数量,该模型具有与原子模拟相当的精度,因此为单分子实验的合理化和涉及 dsDNA 的细胞过程的研究提供了可靠的粗粒度描述。由于其公式的简单性,MADna 可以直接包含在常见的仿真引擎中。特别是,LAMMPS 模型的实现可在在线存储库中获得,以简化其在 DNA 研究社区中的使用。包括拉伸和扭转模量、扭转-拉伸和扭转-弯曲耦合、持续长度和螺距。总体而言,对于检查的数量,该模型具有与原子模拟相当的精度,因此为单分子实验的合理化和涉及 dsDNA 的细胞过程的研究提供了可靠的粗粒度描述。由于其公式的简单性,MADna 可以直接包含在常见的仿真引擎中。特别是,LAMMPS 模型的实现可在在线存储库中获得,以简化其在 DNA 研究社区中的使用。该模型具有与原子模拟相当的精度,因此为单分子实验的合理化和涉及 dsDNA 的细胞过程研究提供了可靠的粗粒度描述。由于其公式的简单性,MADna 可以直接包含在常见的仿真引擎中。特别是,LAMMPS 模型的实现可在在线存储库中获得,以简化其在 DNA 研究社区中的使用。该模型具有与原子模拟相当的精度,因此为单分子实验的合理化和涉及 dsDNA 的细胞过程研究提供了可靠的粗粒度描述。由于其公式的简单性,MADna 可以直接包含在常见的仿真引擎中。特别是,LAMMPS 模型的实现可在在线存储库中获得,以简化其在 DNA 研究社区中的使用。

"点击查看英文标题和摘要"
更新日期:2022-04-08

"点击查看英文标题和摘要"

































 京公网安备 11010802027423号
京公网安备 11010802027423号